修回日期: 2025-10-05
接受日期: 2025-11-14
在线出版日期: 2025-11-28
结肠癌(colon adenocarcinoma, COAD)是常见的肠胃道肿瘤, 发病率与死亡率逐年上升, 需要寻找精确的预后基因来预测患者预后. 在基础研究中发现烯醇化酶3(enolase 3, ENO3)的表达与COAD肿瘤的糖酵解有关.
分析ENO3在结肠癌中的表达和预后情况, 并揭示其在COAD中的潜在分子作用机制.
从癌症基因组图谱(The Cancer Genome Atlas, TCGA)数据库下载COAD表达相关数据, 分析ENO3基因表达情况及其与临床特征的相关性. Kaplan-Meier分析ENO3基因表达和患者预后的关系. 进一步通过COX回归分析评估ENO3基因的独立预后价值. 通过富集分析和免疫浸润分析, 探究ENO3基因在COAD发病中的潜在分子作用.
COAD中的ENO3基因表达水平高于正常结肠组织(P<0.05). COX回归分析发现, ENO3基因是COAD患者的独立预后因子(P<0.05). 功能富集分析结果显示, ENO3基因与物质运输和细胞增殖密切相关. 此外, ENO3基因表达与免疫抑制剂表达相关(P<0.05).
ENO3作为COAD潜在的预后标志物, 参与多种分子调控通路, 可能为免疫治疗提供方向.
核心提要: 烯醇化酶3(enolase 3, ENO3)在结肠癌(colon adenocarcinoma, COAD)中高表达且不利于预后, 可独立预测不良预后及作为治疗COAD的靶点. ENO3通过增强糖酵解可直接促进肿瘤生长, 调节免疫相关基因重塑肿瘤微环境导致免疫抑制, 肿瘤恶化.
引文著录: 张秋颖, 高德, 赵冬梅, 张强, 霍大伟, 李越. ENO3在结肠癌的临床意义及调控机制研究. 世界华人消化杂志 2025; 33(11): 926-936
Revised: October 5, 2025
Accepted: November 14, 2025
Published online: November 28, 2025
Colon cancer (COAD) is a common gastrointestinal tumor, with its incidence and mortality rates rising year by year. There is a need to identify precise prognostic genes to predict patient outcomes. Basic research has found that ENO3 (enolase 3) expression is associated with glycolysis in COAD tumors.
To explore the expression and prognostic value of ENO3 in colon cancer, and to clarify its potential regulatory mechanisms.
COAD related data were downloaded from the Cancer Genome Atlas database to analyze the expression of ENO3 gene and its correlation with clinical features. Kaplan-Meier analysis was performed to assess the relationship between ENO3 gene expression and patient prognosis. The independent prognostic value of ENO3 gene was evaluated through COX regression analysis. The potential mechanism of ENO3 gene in COAD was clarified through enrichment analysis and immune infiltration analysis.
The expression level of ENO3 gene in COAD was higher than that in normal colon tissue (P < 0.05). COX regression analysis revealed that ENO3 gene is an independent prognostic factor for COAD patients (P < 0.05). Functional enrichment analysis showed that the ENO3 gene is closely related to material transport and cell proliferation. In addition, the expression of ENO3 gene was correlated with the expression of immunosuppressants (P < 0.05).
ENO3, as a potential prognostic marker for COAD, participates in various molecular regulatory pathways. This finding may provide a direction for immunotherapy of COAD.
- Citation: Zhang QY, Gao D, Zhao DM, Zhang Q, Huo DW, Li Y. Clinical significance and regulatory mechanism of ENO3 in colon cancer. Shijie Huaren Xiaohua Zazhi 2025; 33(11): 926-936
- URL: https://www.wjgnet.com/1009-3079/full/v33/i11/926.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v33.i11.926
结肠癌(colon adenocarcinoma, COAD)是最常见的胃肠道恶性肿瘤, 其发病率全球排位第3, 是当今沉重的健康负担. 近年来, COAD在全球范围内的发病率仍持续增长[1]. 烯醇酶(enolase, ENO)是由Lohman和Mayerhof于1934年在研究肌肉提取物中3-磷酸甘油酸转化为丙酮酸的过程中发现的[2]. ENO3是ENO基因家族的成员之一, 该基因编码的蛋白在糖酵解过程中发挥重要作用, 并且在不同癌症中异常表达[3]. 目前, 对于ENO3基因在COAD的研究主要集中在该基因与糖酵解途径的关系, 而缺少对该基因在肿瘤的发生、发展、临床病理特征和免疫方面的研究[4]. 因此, 本研究利用公共数据库中有关COAD的基因表达及相关临床数据来分析ENO3基因在COAD中的表达情况, 评估该基因与COAD患者临床病理特征的关系, 并探索其预后价值. 此外, 通过富集分析探索潜在的调控机制, 分析该基因与免疫抑制剂的相关性, 以期为COAD的预防和治疗提供新的生物标志物.
数据来源: 癌症基因组图谱(The Cancer Genome Atlas, TCGA)是美国国家癌症研究所和美国人类基因组研究所共同监督的一个项目. 首先, 使用UCSC Xena(https://xenabrowers.net/datapage/)下载TCGA-COAD的RNAseq数据及相关临床病理信息, 如年龄、性别、病理分期和T分期等. 排除临床信息及表达信息不全的样本, 研究最终纳入474例患者. 根据ENO3基因的中位表达值, 将患者分为高表达组和低表达组. 许多研究结果表明T分期(肿瘤大小和位置)与COAD的进展密切相关, 因此本研究将T分期纳入分析[5,6]. 值得注意的是, TCGA中的T分期和临床分期是根据AJCC癌症分期指南划分的. 具体而言, T1: 肿瘤侵犯黏膜下层; T2: 肿瘤侵犯固有肌层; T3: 肿瘤穿透固有肌层到达结直肠旁组织; T4: 肿瘤侵犯脏层腹膜, 或侵犯邻近器官或结构. 临床分期(S)是按照TNM分期进一步划分的, 具体划分见补充材料. 其次, 使用人类蛋白图谱(https://www.proteinatlas.org)研究ENO3蛋白在正常和COAD组织中的表达情况. 此外, 采用基因表达综合数据库(Gene Expression Omnibus, GEO, https://www.ncbi.nlm.nih.gov/gds)中的GSE39582数据集进行验证.
预后分析: 利用Kaplan-Meier曲线呈现ENO3基因表达水平与COAD患者预后的关系. 使用COX回归分析得出与COAD患者预后相关的独立因子, 性别男、年龄≤60、病理分期S1和T分期T1作为参考水平. 基于多因素COX回归结果, 构建列线图预测COAD患者生存情况. 创建Harrell一致性指数和校准图, 以评估列线图可靠性和准确性.
Limma(Linear models for microarray data)是一种基于广义线性模型的差异表达筛选方法. 使用R软件包Limma(Version 3.40.6)进行差异分析, 获得ENO3基因高低表达两组之间的显著差异基因. 阈值为|Log2(Fold change)|>1.5和P<0.05. 结果采用火山图和热图呈现.
基因本体论(gene ontology, GO)分析、京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes, KEGG)通路分析和基因集富集分析(gene set enrichment anaiysis, GSEA), 均采用R软件包ClusterProfiler(Version 3.14.3). 通过GO分析研究差异基因的细胞组分、分子功能及生物过程. 同时对差异基因可能涉及的KEGG信号通路进行分析. 最后利用GSEA进一步研究ENO3基因潜在的调控机制. P<0.05和发现错误率<0.1认为具有统计学意义.
利用TISIDB(cis.hku.hk/TISIDB/index.php)平台, 挖掘ENO3基因表达与免疫抑制剂的相关性. 用Spearman方法分析免疫检查点抑制剂与ENO3基因表达的相关性. P<0.05认为具有统计学意义.
统计学处理 生物信息学分析及绘图使用R版本3.6.3, 统计学分析使用 SPSS 25.0软件. 采用受试者工作特征(receiver operating characteristic, ROC)曲线判断ENO3基因在COAD中的诊断价值, 并计算曲线下面积(area under the curve, AUC). 采用K-S拟合优度检验处理连续性数据. 对符合正态分布的数据以平均值±标准误差表示, 组间比较采用Student-t检验或对方差分析(analysis of variance, ANOVA); 对不符合正态分布的数据,通过四分位数(Q1-Q4)表示, 并通过秩和检验分析组间差异. 卡方检验分析各临床特征中COAD患者在ENO3基因高低表达两组的分布情况. P<0.05认为具有统计学意义.
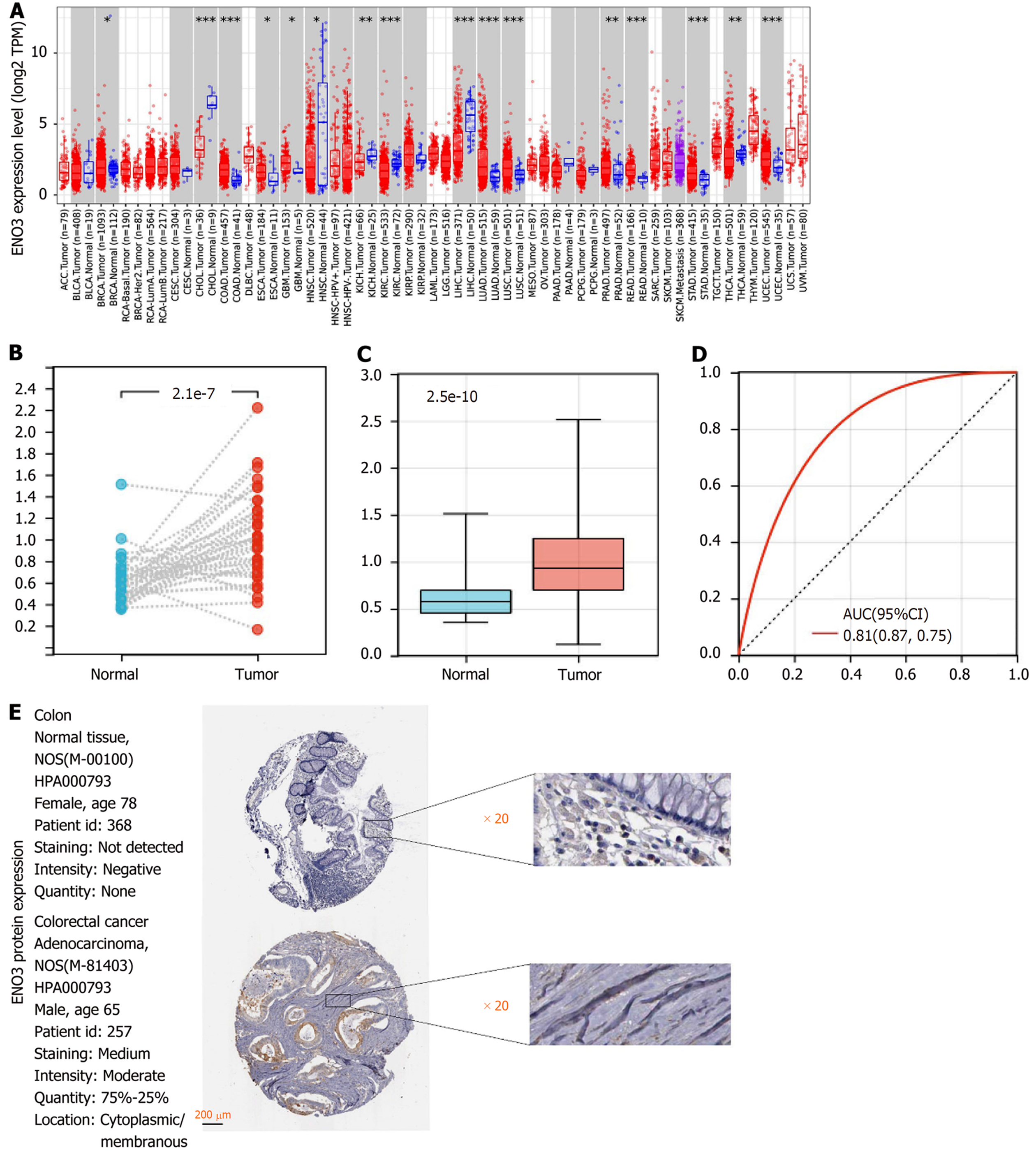
ENO3基因在不同癌种中的表达情况见图1A, ENO3基因在大多数癌种中都显著差异表达, 并在COAD中表达水平显著上调(P<0.001). 利用TCGA-COAD数据做配对和非配对样本分析, 结果均显示COAD中ENO3基因表达水平显著高于正常组织(P<0.05)(图1B和C). ROC曲线结果表明ENO3基因表达水平对COAD有较高的诊断价值(AUC = 0.81)(图1D). 通过对ENO3的免疫组织化学分析发现COAD组织呈现阳性而正常组织呈阴性(图1E).
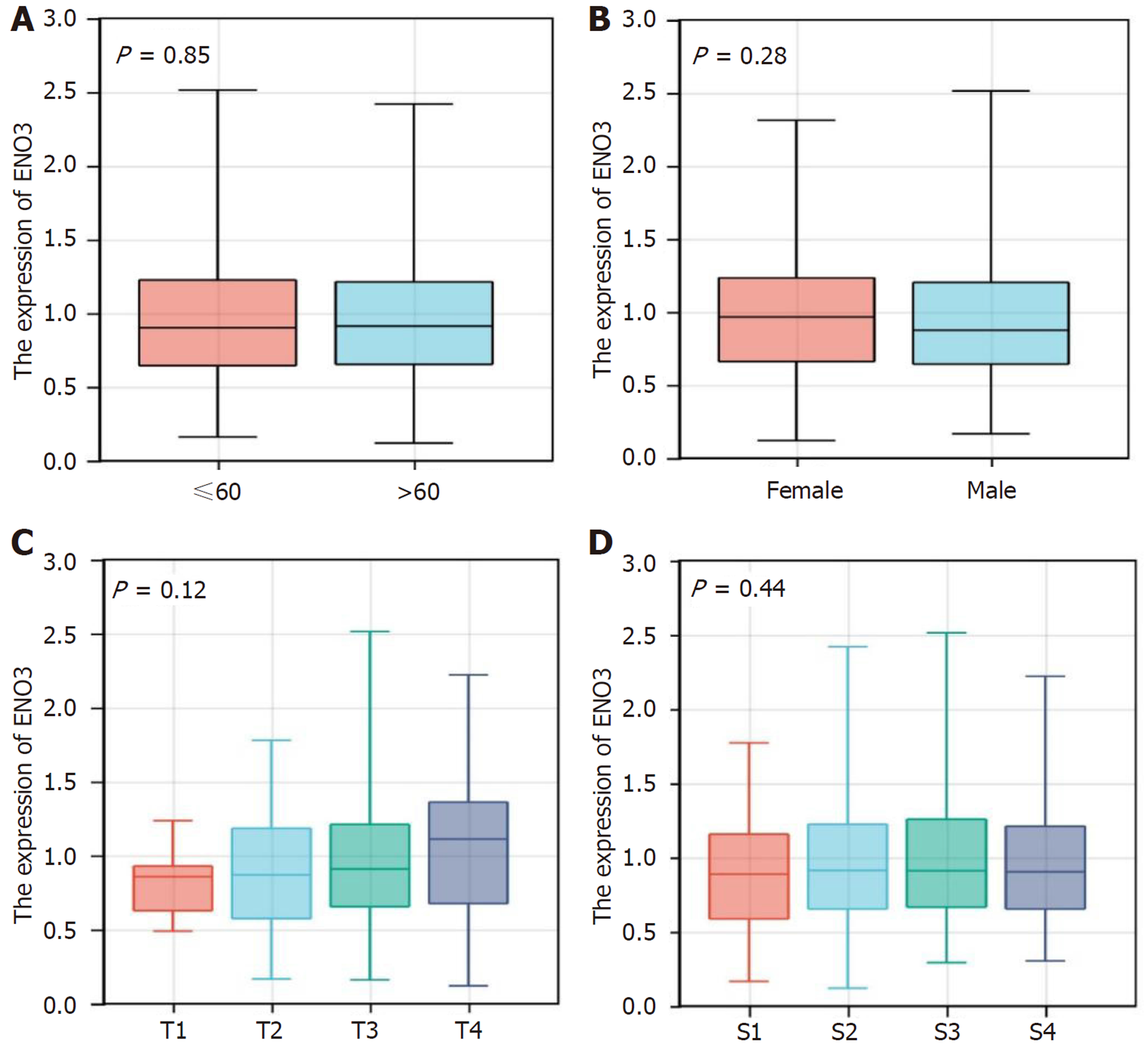
COAD患者根据ENO3表达水平的中位数(0.9059)将患者分为ENO3高表达组, ENO3低表达组, 进而比两组的临床特征. 卡方检验结果显示ENO3基因表达与病理分期、性别、T分期和年龄无关, 差异无统计学意义(P>0.05)(表1). 进一步ANOVA和秩和检验结果显示ENO3基因表达与COAD的各临床特征相关性无统计学意义(P>0.05)(图2).
| 临床特征 | ENO3表达[n(%)] | χ2 | P值 | |
| 低表达(n = 237) | 高表达(n = 237) | |||
| 病理分期 | 0.832 | 0.842 | ||
| S1 | 43(9.1) | 36(7.6) | ||
| S2 | 94(19.8) | 99(20.9) | ||
| S3 | 65(13.7) | 68(14.3) | ||
| S4 | 35(7.4) | 34(7.2) | ||
| 性别 | 2.742 | 0.098 | ||
| 女 | 103(21.7) | 121(25.5) | ||
| 男 | 134(28.3) | 116(24.5) | ||
| T分期 | 3.214 | 0.360 | ||
| T1 | 6(1.3) | 4(1) | ||
| T2 | 44(9.3) | 35(7.4) | ||
| T3 | 164(34.6) | 165(34.8) | ||
| T4 | 23(4.9) | 33(7.0) | ||
| 年龄 | 0.250 | 0.617 | ||
| ≤60 | 74(15.6) | 69(14.6) | ||
| >60 | 163(34.4) | 168(35.4) | ||
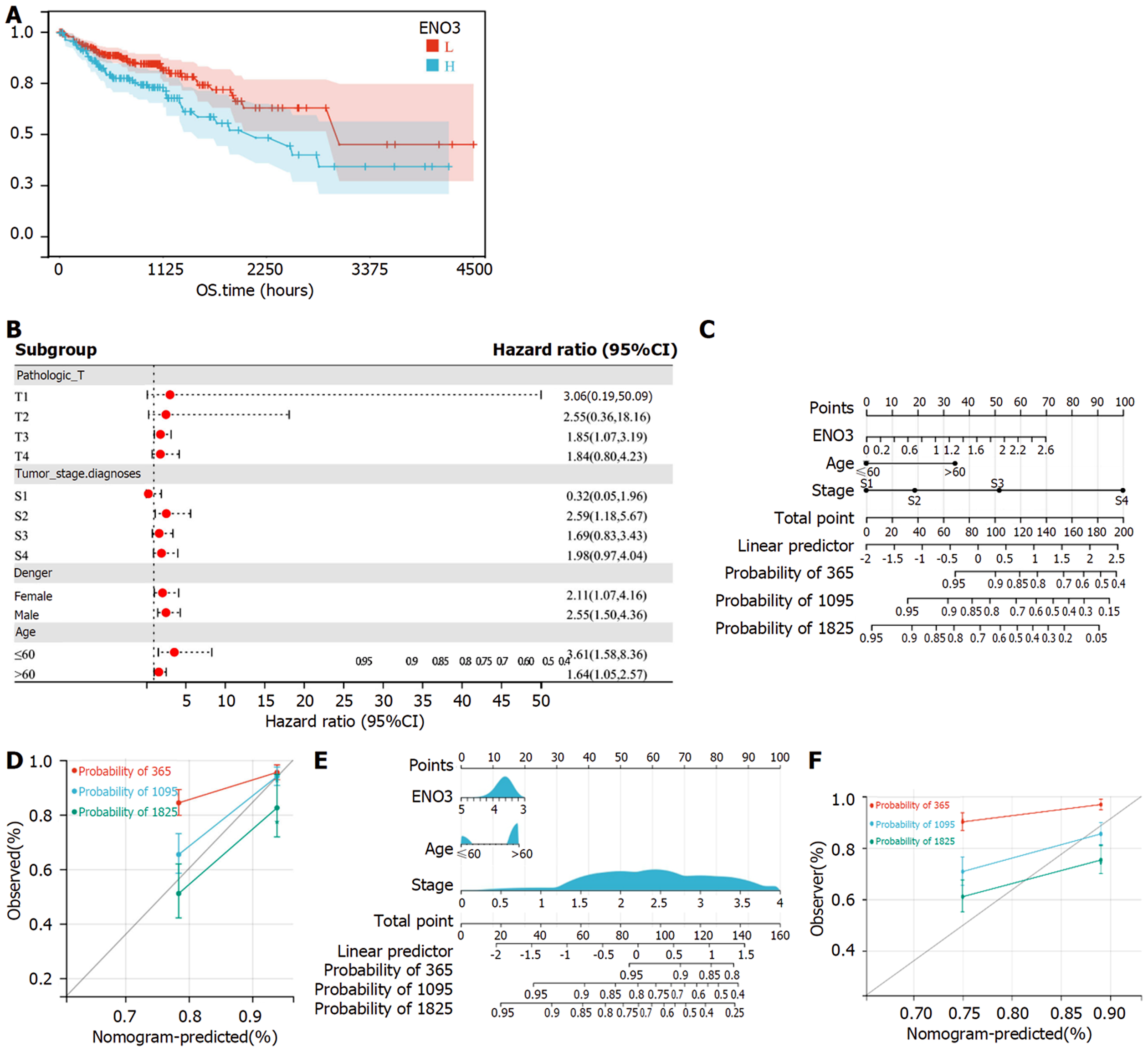
相较于ENO3基因低表达组患者, ENO3基因高表达组患者的总生存率更低(P<0.05)(图3A). 此外, 按照临床参数将COAD样本分为不同的亚组, 探究各个亚组中ENO3基因表达水平与预后的关系, 发现在T分期T3[HR(95%CI): 1.85(1.07-3.19), P<0.05]和病理分期S2[HR(95%CI): 2.59(1.18-5.67), P<0.05]亚组中ENO3基因高表达与不良预后相关, 差异有统计学意义(图3B).
为了探究ENO3基因的独立预后价值, 单变量COX分析结果显示T分期、病理分期和ENO3基因都是COAD患者重要的预后因子(P<0.05)(表2). 将P<0.1的因素纳入多因素COX分析中, 结果显示年龄、病理分期和ENO3基因都是COAD患者的独立预后因子(P<0.05).
| 临床特征 | 数量(N) | 单因素分析 | 多因素分析 | ||
| HR(95%CI) | P值 | HR(95%CI) | P值 | ||
| 性别 | 474 | 0.847(0.573-1.253) | 0.407 | ||
| T分期 | 474 | 2.808(1.885-4.183) | <0.001 | 1.588(0.983-2.565) | 0.059 |
| 病理分期 | 474 | 2.178(1.738-2.730) | <0.001 | 2.174(1.680-2.813) | <0.001 |
| 年龄 | 474 | 1.559(0.979-2.483) | 0.061 | 2.149(1.335-3.460) | <0.001 |
| ENO3 | 474 | 1.747(1.126-2.711) | 0.013 | 1.648(1.056-2.573) | 0.028 |
由独立预后因子组成的列线图, 结果显示ENO3基因对COAD患者的预后生存率起重要作用(图3C). 模型总体的一致性指数为0.738, 说明列线图的预测能力良好(图3D). 并在GSE39582数据集进行了验证, 结果显示模型总体在验证集中一致性为0.644(图3E和F). 所有结果都表明在COAD患者中ENO3基因表达增加与预后不良有关.
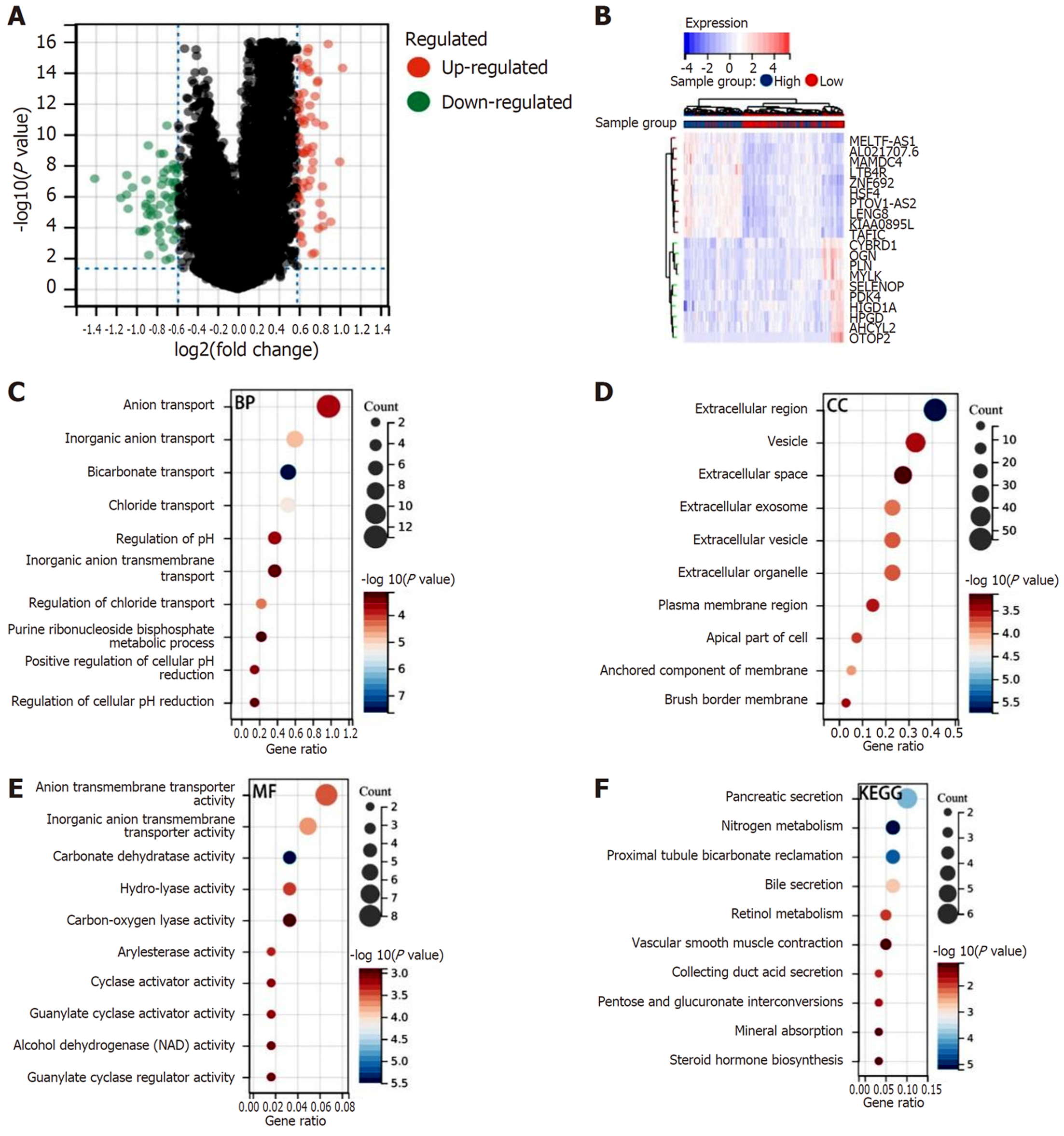
利用Limma方法筛选ENO3基因高低两组之间的差异基因, 结果有183个显著差异表达基因, 其中有125个上调基因和58个下调基因(图4A). 使用热图显示前10上调基因和前10下调基因(图4B). 采用GO和KEGG通路分析探索差异基因的潜在机制. 通过差异基因的功能注释后, 分析出差异基因主要集中在物质运输和酶促反应, 如阴离子跨膜转运、无机阴离子跨膜转运蛋白、水解酶活性和碳氧裂解酶活性等, 以及一些生物学过程, 如阴离子运输、氢化物运输、pH调节和氯化物运输管制等(图4C-E). 通过差异基因的通路富集, 我们发现主要集中在小肠消化液分泌通路, 如胰液分泌、胆汁分泌和收集导管酸分泌等(图4F).
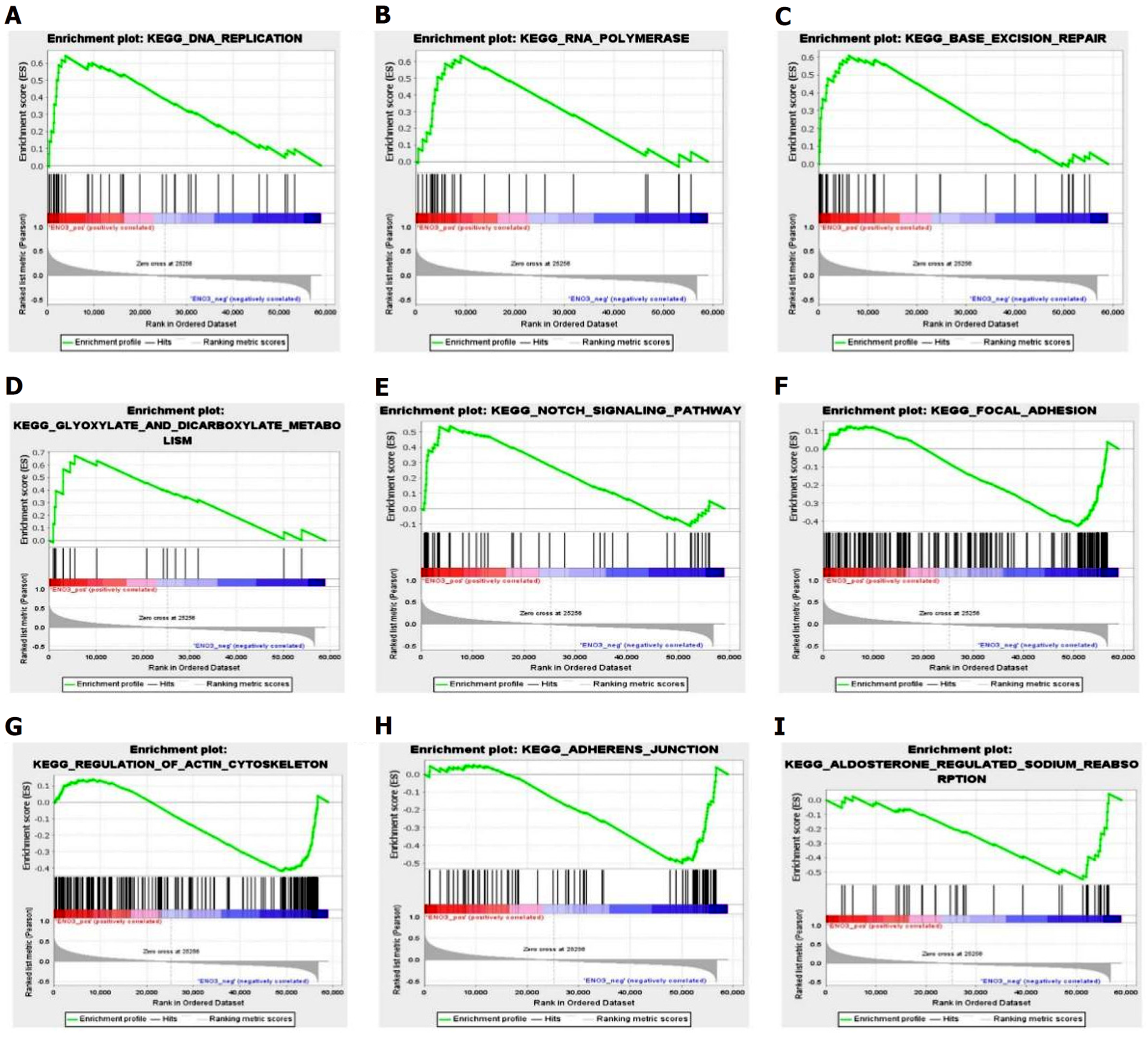
此外, 利用GSEA挖掘ENO3基因在COAD相关的机制, 我们发现主要集中在肿瘤相关信号通路, 与DNA复制、RNA聚合酶、碱基切除修复、乙醛酸盐和二羧酸盐代谢和Notch信号通路呈正相关(P<0.05)(图5A-E), 与附着斑、黏合连接、机动蛋白细胞骨架调节和醛固酮调节的钠重吸收呈负相关(P<0.05)(图5F-I).
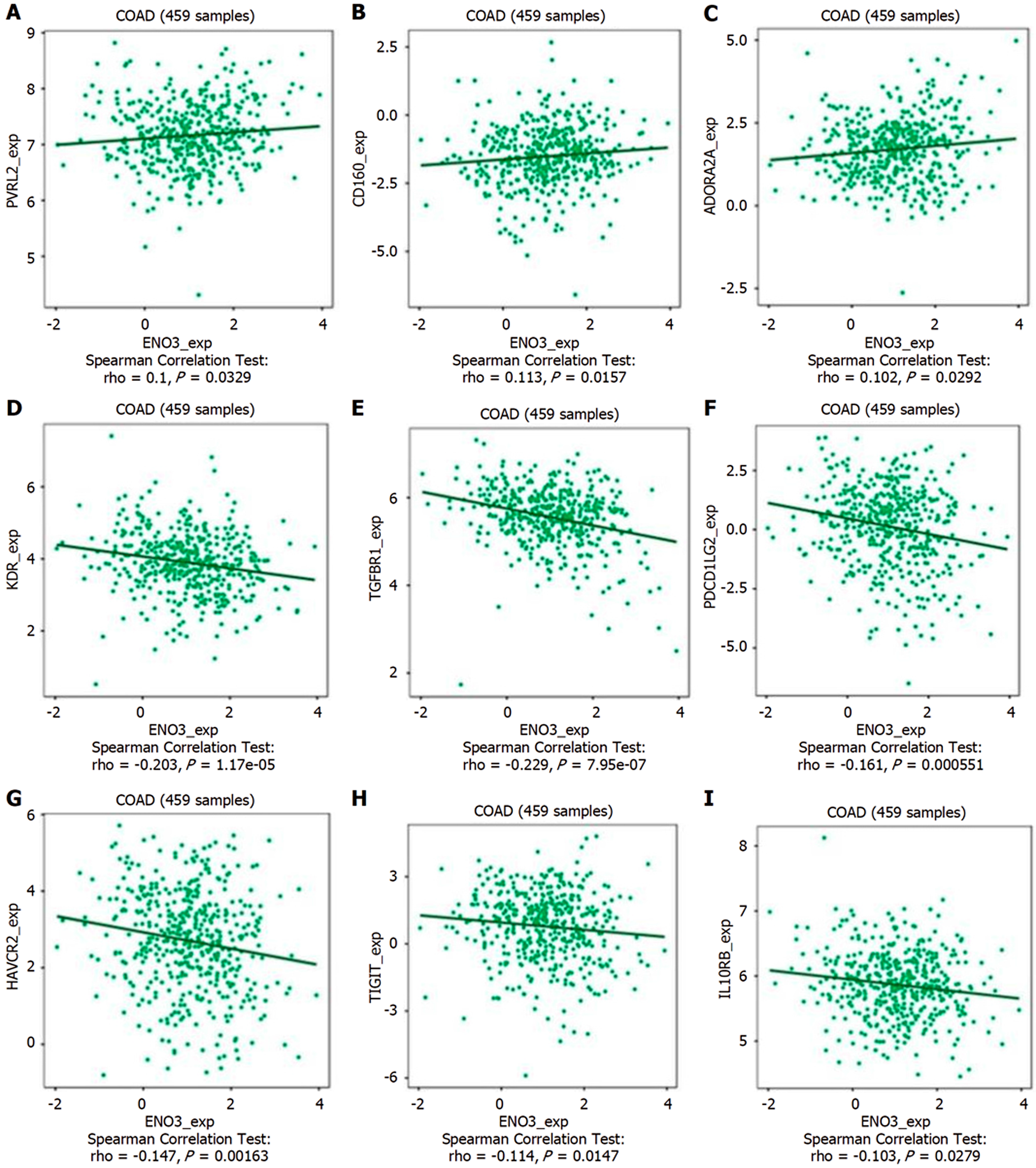
采用TISIDB平台分析ENO3基因表达水平与免疫检查点之间的相关性, 发现ENO3基因与PVRL2、CD160和ADORA2A呈正相关(P<0.05)(图6A-C), 与KDR、TGFBR1、PDCD1LG2、HAVCR2、TIGIT和IL10RB呈负相关(P<0.05), 差异均有统计学意义(图6D-I).
COAD是一种普遍且高致死率的恶性肿瘤, 其发病率呈逐年上升的趋势. 有研究表明, 早期COAD患者生存率可达到90%, 但晚期患者的生存率仅15%[7]. 癌症早期诊断主要依靠肿瘤标志物进行筛选和诊断, 但COAD常规肿瘤标志物, 如CEA、CA199和CA195等, 都不是COAD特异的肿瘤标志物[8,9]. 因此, 探索新的COAD肿瘤标志物对COAD的早期诊断和靶向治疗显得至关重要.
ENO3基因为烯醇化酶家族的一员, 通过调控糖酵解途径和催化2-磷酸甘油过程在细胞中发挥作用[10]. 多项研究表明[3,11], ENO3基因在肝癌、胃癌和肾癌的进展中起重要作用. 然而, ENO3基因在COAD作用机制仍未阐明.
本研究使用TCGA数据库获得了474例COAD患者数据, 通过COAD组织和正常组织ENO3基因表达的比较, 以及ENO3基因与临床特征关系的分析, 得出ENO3基因在COAD组织中高表达且与临床特征相关性无统计学意义. 我们推断ENO3基因高表达可能不受上述临床特征的影响而促进COAD的进展. 同时, 通过ENO3基因在COAD中的高诊断效能, 可以推测出ENO3基因可能是COAD潜在的诊断标志物. 综合COX回归分析和列线图的结果, 表明ENO3基因在COAD患者的预后中发挥重要作用.
该研究发现的ENO3在COAD中高表达且不利于预后与多数研究一致. 如Mao等[12]研究在基于糖酵解和缺氧联合基因构建COAD预后预测模型时, 也发现ENO3在肿瘤中高表达[12]. Pan等[13]研究表明在结肠癌中, ENO3是高表达, 且与不良预后相关[13]. 该研究中发现高低ENO3表达分组的差异表达基因也富集在糖酵解途径, 说明ENO3基因参与了肿瘤细胞的糖酵解过程; 通过ENO3基因的GSEA分析发现, ENO3基因在COAD中与黏着斑、Notch信号通路和DNA复制等经典的肿瘤相关信号通路密切相关. 这些信号通路直接或间接调控细胞活动, 如黏附、迁移、分化、增殖和细胞凋亡[12-14]. 这些结果提示ENO3基因参与了COAD发展, 与已报道的ENO3通过增强糖酵解从而促进结肠癌的发展相符. 已报道研究[10]通过实验发现上调COAD细胞中的ENO3基因可提高乳酸和ATP的产量, 下调ENO3基因, 可得到相反的效应, 证明了ENO3在COAD中可以增强线粒体外糖酵解过程. 此外, 该研究还通过克隆形成, 划痕等细胞恶性表型实验, 发现ENO3的上调促进肿瘤恶性表型发展, 说明ENO3上调增加糖酵解为肿瘤发展提供了能量.
最近, 孟凡来等[14]通过免疫组化染色技术发现ENO3蛋白高表达不有利于COAD患者的总生存期, 但是与肿瘤特征(如病理分期, 肿瘤大小等)无关, 提出ENO3在结COAD中的生物学作用可能并非通过糖酵解途径促进结直肠癌发生发展, 而是与治疗反应有关的观点. 而在我们的研究发现尽管ENO3的表达水平在不同T分期和S分期中无显著差异, 但是仅在T3和S2患者中, 显著与患者的预后有关. 因此, 不能赞同孟凡来等[14]研究中非通过糖酵解途径促进肿瘤发展的观点. 我们整合本研究的结果与已报道的研究结论认为, ENO3通过糖酵解促进COAD发展是其影响患者生存的基础, 而这个过程同时受其他因素如患者的免疫反应能力, 治疗情况等影响, 因此, 需要深入研究.
在癌症患者中, 免疫检查点抑制剂通过抑制检查点途径来增强患者的免疫反应[15]. 该研究通过对COAD中的ENO3基因的表达与免疫抑制检查点的探索, 发现ENO3基因可能调控PVRL2诱导并抑制CD8+T细胞功能, 调控CD160控制NK细胞产生细胞因子, 调控PDCD1GL2影响第三淋巴组织的发育等[16-18]. 此外, ENO3作为糖酵解酶, 其高活性会驱动Warburg效应, 导致大量乳酸的产生和堆积[11], 进而上调CD39/CD73 ectoenzymes的表达, 导致细胞外腺苷水平升高. CD39/CD73是催化ATP逐步降解为腺苷的关键酶. ADORA2A是腺苷A2A受体, 可激活腺苷酸环化酶, 并提高细胞内免疫抑制性cAMP的水平, 腺苷信号在肿瘤微环境中可抑制T细胞功能和促进免疫耐受[19,20]. ENO3与ADORA2A呈正相关关系表明ENO3高表达可能通过这个途径增强免疫抑制状态. 总之, ENO3基因可能通过调节免疫检查点抑制剂促进COAD的进展, 这为ENO3基因成为COAD诊断及治疗的生物标记物提供了理论依据.
此外, 该研究基于ENO3基因表达水平与重要的临床特征构建的预后预测列线图具有良好的预测效能, 对临床上预测患者的1, 3, 5年生存概率具有重要参考价值. 但是本研究也存在一定的局限性. 第一, 研究主要是依赖于生物信息学分析, 用于研究的数据大多数都是基于公共数据库中挖掘的数据, 需要进一步的实验验证来阐明这些预测的生物功能是否在COAD的发展中发挥作用, 从而可以让我们进一步了解COAD中的ENO3基因; 第二, 研究的数据库数量有限, 我们应该在多个数据库中进一步验证我们的发现. 因此, 需要大样本量的前瞻性研究来验证本研究的结论.
综上所述, ENO3在结肠癌中可能通过双重机制发挥作用: 一方面, 通过增强糖酵解直接促进肿瘤生长; 另一方面, 通过调节免疫相关基因重塑肿瘤微环境, 导致免疫抑制和不良预后. 这些发现提示ENO3可作为COAD的潜在生物标志物和治疗靶点.
结肠癌(colon adenocarcinoma, COAD)发病率与死亡率逐年上升, 尽管有多种基因被发现可预测患者不良预后, 但是效果总是差强人意. 烯醇化酶3(enolase 3, ENO3)作为调控糖酵解途径的关键基因, 已经被发现其与COAD有关, 是COAD潜在的预测因子, 然而其在COAD的发展及不良预后预测中的作用不清晰. 因此, 探究他们间的关系, 分析ENO3作为COAD的预后因子的可能性, 为补充ENO3在COAD中的作用及预测COAD不良预后价值提供参考.
本研究先探究了ENO3在COAD中的表达水平, 然后探究ENO3基因与COAD患者的临床特性包含年龄, 性别, 病理分期, T分期间的关系. 通过生物信息学分析探究ENO3在COAD发生中的作用机制. 该研究拟解决了ENO3在COAD中的作用机制不清晰的问题, 以及其是否可以作为COAD不良预后的预测因子的问题. 本研究在补充ENO3在肿瘤中的作用机制知识和在开发新的COAD不良预后的预测因子上具有十分重要的意义.
本研究的主要目标是探究ENO3的表达与COAD的临床特性之间关系, 其是否可为不良预后预测因子, 及ENO3在影响COAD发展的潜在机制. 最终本研究实现了明确ENO3的表达与患者不良预后之间的关系. 且筛选了ENO3在影响COAD发展参与的通路及阐述了可能的生物学机制. 该研究在推动ENO3在预测COAD不良预后的临床应用具有重要意义.
本研究采用包含Kaplan-Meier曲线, COX生存分析, 差异基因表达分析, 基因富集分析等生物信息学分析方法在癌症基因组图谱(The Cancer Genome Atlas, TCGA)数据库中进行了ENO3的表达与COAD预后间关系的探究, 且在基因表达综合数据库(Gene Expression Omnibus, GEO)数据集中进行了验证. 且采用数据统计分析对ENO3表达与临床特征间的关系进行了探究. 这些研究方法在探究单基因在肿瘤中的作用及预测价值提供了一个创新范例. 具体而言, 从基因表达水平-基因与临床特征间关系-潜在机制探索形成一个完整的探究体系. 其次, 该研究充分利用公共数据库TCGA数据集探究和GEO数据集验证, 增加了研究结论的可靠度.
经过本研究的数据分析, 发现ENO3高表达有利于COAD患者的不良预后, 结合临床特征对预测患者预后具有良好效能. 通过生物信息学探究发现这个过程参与多条通路如DNA复制, 碱基切除修复等. 本研究结果在探究ENO3作为COAD新的预后预测因子的领域中提供了理论依据.
本研究发现ENO3在COAD中高表达, 通过增强糖酵解和调控免疫微环境促进肿瘤发展, 且不利于患者预后; 提出作为COAD患者的不良预后预测因子新理论. 本研究的结论对ENO3与COAD肿瘤相关性的促肿瘤方向进行了确定性归纳. 在ENO3促进肿瘤机制提出了调控免疫相关基因重塑肿瘤微环境的独到见解. 提出ENO3不仅通过增强糖酵解促进肿瘤发展的假说. 提出ENO3基因与免疫检查点相关性分析方法和ENO3表达联合临床特征构建预后模型的新方法. 通过数据分析发现, 基因富集分析ENO3与多种肿瘤发展相关通路相关, ENO3与一些免疫检查点相关的新现象, 同时也证实了我们提出的假说. 该研究基于ENO3和临床特征构建的列线图可以用于临床实践上预测COAD的1, 3, 5年生存期概率.
本研究的研究模式为在多组学上研究单基因在肿瘤中的作用及临床价值提供了经验. 本研究未来研究的方向是通过体内外实验深入探究ENO3通过双重机制促COAD发展中的详细细节.
| 1. | Wagle NS, Nogueira L, Devasia TP, Mariotto AB, Yabroff KR, Islami F, Jemal A, Alteri R, Ganz PA, Siegel RL. Cancer treatment and survivorship statistics, 2025. CA Cancer J Clin. 2025;75:308-340. [PubMed] [DOI] |
| 2. | Pancholi V. Multifunctional alpha-enolase: its role in diseases. Cell Mol Life Sci. 2001;58:902-920. [PubMed] [DOI] |
| 3. | Cui H, Guo D, Zhang X, Zhu Y, Wang Z, Jin Y, Guo W, Zhang S. ENO3 Inhibits Growth and Metastasis of Hepatocellular Carcinoma via Wnt/β-Catenin Signaling Pathway. Front Cell Dev Biol. 2021;9:797102. [PubMed] [DOI] |
| 4. | Subramanian A, Miller DM. Structural analysis of alpha-enolase. Mapping the functional domains involved in down-regulation of the c-myc protooncogene. J Biol Chem. 2000;275:5958-5965. [PubMed] [DOI] |
| 5. | Zheng C, Xu L, Ou B, Abdourahaman IMB, Chen X, Xu H, Zheng Y, Pan Y. Prognostic significance of tumor deposit counts in stage III colorectal cancer based on T/N staging and chemotherapy status: A retrospective cohort study. Surg Open Sci. 2025;24:42-50. [PubMed] [DOI] |
| 6. | Turan B, Sanli AN, Sanli DET, Acar S. Prognostic impact and independent significance of tumor deposits in early-stage colon cancer: a population-based cohort study. Int J Colorectal Dis. 2025;40:146. [PubMed] [DOI] |
| 7. | Mattiuzzi C, Sanchis-Gomar F, Lippi G. Concise update on colorectal cancer epidemiology. Ann Transl Med. 2019;7:609. [PubMed] [DOI] |
| 8. | Teijeira A, Migueliz I, Garasa S, Karanikas V, Luri C, Cirella A, Olivera I, Cañamero M, Alvarez M, Ochoa MC, Rouzaut A, Rodriguez-Ruiz ME, Sanmamed MF, Klein C, Umaña P, Ponz M, Bacac M, Melero I. Three-dimensional colon cancer organoids model the response to CEA-CD3 T-cell engagers. Theranostics. 2022;12:1373-1387. [PubMed] [DOI] |
| 9. | Yang Y, Meng WJ, Wang ZQ. MicroRNAs in Colon and Rectal Cancer - Novel Biomarkers from Diagnosis to Therapy. Endocr Metab Immune Disord Drug Targets. 2020;20:1211-1226. [PubMed] [DOI] |
| 10. | Chen J, Zhang Z, Ni J, Sun J, Ju F, Wang Z, Wang L, Xue M. ENO3 promotes colorectal cancer progression by enhancing cell glycolysis. Med Oncol. 2022;39:80. [PubMed] [DOI] |
| 11. | Wang J, Ju HJ, Zhang F, Tian H, Wang WG, Ma YL, Xu WS, Wang YH. A novel NSUN5/ENO3 pathway promotes the Warburg effect and cell growth in clear cell renal cell carcinoma by 5-methylcytosine-stabilized ENO3 mRNA. Am J Transl Res. 2023;15:878-895. [PubMed] |
| 12. | Mao G, Wu J, Cui H, Dai L, Ma L, Zhou Z, Liang B, Zhang S, Lin S. A Novel Glycolysis and Hypoxia Combined Gene Signature Predicts the Prognosis and Affects Immune Infiltration of Patients with Colon Cancer. Int J Gen Med. 2022;15:1413-1427. [PubMed] [DOI] |
| 13. | Pan X, Wu H, Chen G, Li W. Prognostic Value of Enolase Gene Family in Colon Cancer. Med Sci Monit. 2020;26:e922980. [PubMed] [DOI] |
| 14. | 孟 凡来, 梁 晓东. 烯醇化酶3表达与结直肠癌临床病理特征及生存相关性分析. 湖北科技学院学报(医学版). 2024;38:519-22, 30. [DOI] |
| 15. | Bruera S, Suarez-Almazor ME. The effects of glucocorticoids and immunosuppressants on cancer outcomes in checkpoint inhibitor therapy. Front Oncol. 2022;12:928390. [PubMed] [DOI] |
| 16. | Whelan S, Ophir E, Kotturi MF, Levy O, Ganguly S, Leung L, Vaknin I, Kumar S, Dassa L, Hansen K, Bernados D, Murter B, Soni A, Taube JM, Fader AN, Wang TL, Shih IM, White M, Pardoll DM, Liang SC. PVRIG and PVRL2 Are Induced in Cancer and Inhibit CD8(+) T-cell Function. Cancer Immunol Res. 2019;7:257-268. [PubMed] [DOI] |
| 17. | Tu TC, Brown NK, Kim TJ, Wroblewska J, Yang X, Guo X, Lee SH, Kumar V, Lee KM, Fu YX. CD160 is essential for NK-mediated IFN-γ production. J Exp Med. 2015;212:415-429. [PubMed] [DOI] |
| 18. | Masugi Y, Nishihara R, Hamada T, Song M, da Silva A, Kosumi K, Gu M, Shi Y, Li W, Liu L, Nevo D, Inamura K, Cao Y, Liao X, Nosho K, Chan AT, Giannakis M, Bass AJ, Hodi FS, Freeman GJ, Rodig SJ, Fuchs CS, Qian ZR, Nowak JA, Ogino S. Tumor PDCD1LG2 (PD-L2) Expression and the Lymphocytic Reaction to Colorectal Cancer. Cancer Immunol Res. 2017;5:1046-1055. [PubMed] [DOI] |
| 19. | Zhang C, Wang K, Wang H. Adenosine in cancer immunotherapy: Taking off on a new plane. Biochim Biophys Acta Rev Cancer. 2023;1878:189005. [PubMed] [DOI] |
| 20. | Chen S, Xu Y, Zhuo W, Zhang L. The emerging role of lactate in tumor microenvironment and its clinical relevance. Cancer Lett. 2024;590:216837. [PubMed] [DOI] |
学科分类: 胃肠病学和肝病学
手稿来源地: 宁夏回族自治区
同行评议报告学术质量分类
A级 (优秀): 0
B级 (非常好): 0
C级 (良好): C, C
D级 (一般): D, D
E级 (差): 0
科学编辑: 刘继红 制作编辑:张砚梁