修回日期: 2025-10-04
接受日期: 2025-10-15
在线出版日期: 2025-10-28
结直肠癌(colorectal cancer, CRC)是全球第三高发恶性肿瘤, 其不良预后主要归因于肿瘤转移和基因突变等因素. 在我国, CRC发病率亦呈逐年升高趋势, 疾病负担日益严. 单细胞测序技术(single-cell sequencing, SCS)可在单个细胞分辨率下实现基因组、转录组和表观遗传学分析, 揭示肿瘤转移的分子机制及肿瘤微环境的复杂动态特征, 在CRC研究中发挥重要作用. 该技术有助于阐明肿瘤的发生、发展与转移的驱动机制, 为肿瘤细胞异质性、循环肿瘤细胞和肿瘤药物靶点的研究提供新视角. SCS技术在多组学整合及临床转化中的潜力巨大, 为转移机制研究、靶点发现和个性化诊疗策略提供了重要支持. 本文综述了SCS技术的发展、多组学测序及与人工智能带来的挑战.
核心提要: 单细胞测序技术以极高分辨率解析结直肠癌的肿瘤异质性、循环肿瘤细胞及肿瘤微环境动态演变, 揭示细胞亚群功能、转移机制与免疫调控网络. 结合人工智能与多组学整合, 该技术推动精准靶点发现及个体化治疗策略构建, 为结直肠癌研究提供全新视角与临床转化潜力.
引文著录: 宫甜甜, 刘艳坤. 单细胞测序技术在结直肠癌中的研究进展. 世界华人消化杂志 2025; 33(10): 771-780
Revised: October 4, 2025
Accepted: October 15, 2025
Published online: October 28, 2025
Colorectal cancer (CRC) is the third most common malignancy worldwide. The poor prognosis of CRC is primarily driven by tumor metastasis and genetic mutations. In China, the incidence of CRC continues to rise, contributing to an increasing public health burden. Single-cell sequencing (SCS) provides high-resolution genomic, transcriptomic, and epigenetic profiling at the single-cell level. It helps uncover the molecular mechanisms of tumor metastasis and the dynamic complexity of the tumor microenvironment, and plays a critical role in CRC research. It elucidates the processes of tumor initiation, progression, and dissemination, providing new insights into tumor heterogeneity, circulating tumor cells, and therapeutic targets. Moreover, SCS shows great considerable promise in multi-omics integration and clinical translation, supporting studies on metastasis mechanism, target identification, and the development of personalized therapeutic strategies. This review summarizes recent advances in SCS and multi-omics sequencing technologies, as well as the challenges in integrating SCS with artificial intelligence.
- Citation: Gong TT, Liu YK. Advances in application of single-cell sequencing in colorectal cancer. Shijie Huaren Xiaohua Zazhi 2025; 33(10): 771-780
- URL: https://www.wjgnet.com/1009-3079/full/v33/i10/771.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v33.i10.771
结直肠癌(colorectal cancer, CRC)是全球高发且死亡率居高不下的消化道恶性肿瘤之一[1]. 根据最新统计显示CRC是位居肺癌和乳腺癌之后的第三大恶性肿瘤, 且男性患病率大于女性[2]. 对社会经济发展与公共卫生体系构成严峻挑战. 晚期CRC患者的高病死率主要源于肿瘤快速进展和远处转移, 转移性患者5年生存率约为10%[3]. CRC最常见的转移部位是肝脏, 约15%-25%的患者在初诊时已发生肝转移, 且多表现为化疗耐药及预后不良[4]. 肿瘤微环境(tumor microenvironment, TME)在CRC进展中发挥核心调控作用[5], 但TME内多种细胞及其分泌的细胞因子促进CRC发展协同机制尚未完全阐明.
肿瘤异质性是导致CRC治疗困难的核心因素, CRC高度异质性驱动肿瘤细胞适应TME变化, 从而增强侵袭性与耐药性, 这也是现有治疗方案疗效受限的重要原因[6,7]. 传统分析方法仅能分析肿瘤组织整体特征, 难以揭示细胞层面的癌症的发生、发展及转移的内在机制, 从而忽视肿瘤细胞的内在异质性导致错失关键生物信息[8]. 随着分子生物学和测序技术的革新, 单细胞测序(single-cell sequencing, SCS)技术兴起, 为解析细胞异质性与细胞间相互作用提供了单细胞分辨率的全新视角, 精准鉴定少见细胞类型和功能亚群, 突破传统分析方法局限性[9]. 本文将综述SCS技术在CRC中的研究进展.
SCS技术是指在单个细胞水平均匀扩增DNA或RNA, 进行标记建库和高通量测序, 获得单个细胞的基因表达谱, 从而揭示细胞的遗传物质信息. 该技术可深入解析细胞异质性, 并对单个细胞的基因组、转录组和表观遗传组进行全面研究, 为理解细胞功能和状态提供新视角[10-12]. 此外, SCS技术可识别细胞亚群、解析细胞间通讯和调控网络, 有助于揭示疾病发生发展的复杂机制, 并推动疾病预后判断与药物研发.
单细胞研究始于早期分子生物学技术的发展. 自聚合酶链式反应(polymerase chain reaction, PCR)问世, 科学家们便尝试将其用于单细胞系统的精细分析. Sanger第一代基因测序技术出现[13], 使得基因测序成为研究细胞遗传变异与疾病的重要工具. 2005年, 二代基因测序技术技术出现了登上历史舞台, 该技术具有高通量、低成本的优点, 能够一次对几百万条DNA分子进行测序. 但是二代测序无法进行全基因组覆盖, 读长较短. 因此, 出现了第三代测序技术, 该技术通量更高且读取长度(>20 kb)能够有效提高对遗传信息的捕获能力, 不需要扩增DNA就可直接测序[14]. 2009年, Tang等[15]首次利用高通量测序对小鼠卵母细胞和单卵裂球细胞分析, 正式提出SCS概念, 开启了SCS的新纪元[16]. 2011年, SCS技术开始应用于人类肿瘤细胞DNA测序[17]. 2013年, 《Nature Methods》和《Science》均将其评为年度最具创新性的技术之一, 并列为最值得关注的六大领域之首[18]. 随着测序与扩增技术不断进步, 尤其是微流控技术(Drop-seq[19]、inDrop[20]和Microwell-seq[21])的发展, SCS实现了高通量, 打破了早期测序通量低的限制. SCS技术正在重塑生命科学研究格局, 推动肿瘤学、免疫学、植物学等领域展现广阔的临床应用前景[15].
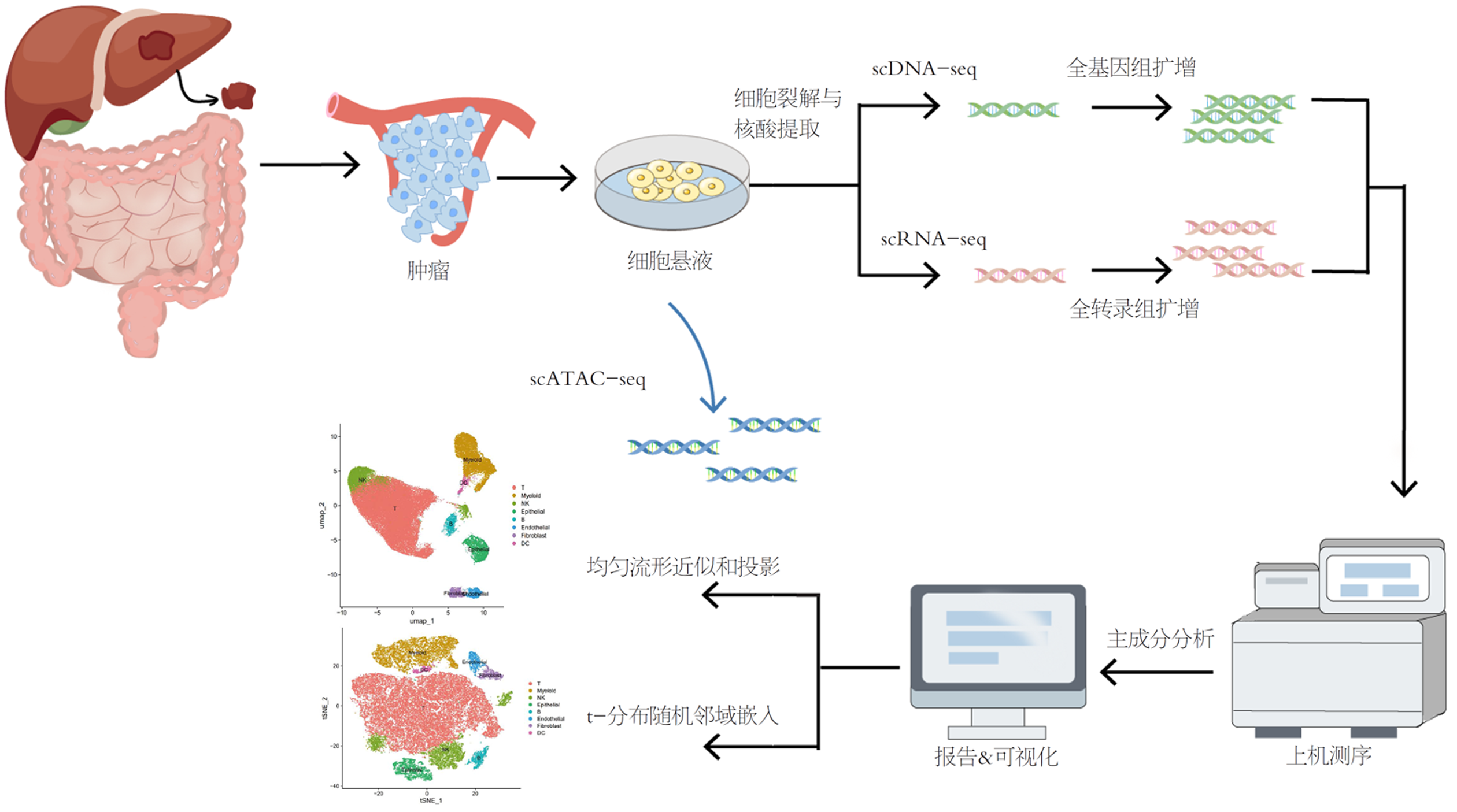
SCS技术包括单细胞DNA测序(single-cell DNA sequencing, scDNA-seq)、单细胞RNA测序(single-cell RNA sequencing, scRNA-seq)和单细胞ATAC测序(single-cell ATAC sequencing, scATAC-seq)等(图1). scDNA-seq的出现极大的推动了基因组学研究, 该技术主要聚焦于DNA分子的异常和变异, 可在单细胞水平捕捉拷贝数变异和单核苷酸多态性[22], 为揭示细胞异质性、分化过程与演化机制提供重要信息. 表1总结了应用最多的scDNA-seq技术[23-27]. 目前, 应用最为广泛的是scRNA-seq技术, 该技术可在单个细胞水平全面捕获转录本, 直观揭示细胞间的异质性, 准确区分细胞在不同状态或发育阶段的特异性分子特征[28], 在揭示基因间调控关系和组织构成方面具有重要作用, 表2总结了当前主要scRNA-seq技术[21,29-34]. 其次, scATAC-seq则聚焦于染色质的开放状态. scATAC-seq利用高活性的Tn5转座酶选择性标记并切割开放的染色质区域, 可深入解析单细胞水平的染色质开放状态和基因调控的表观遗传机制[35]. 通过整合细胞捕获与文库构建, 显著提升了实验效率和通量[35].
| 方法 | 原理 | 优点 | 不足 | 参考文献 |
| 连接锚定型PCR | 用蛋白酶K处理单细胞释放DNA, 用限制性内切酶将DNA切割成片段, 链接特异接头形成起始模版, 变性、退火和延伸进行循环扩增 | 高精度检测DNA拷贝数变异和基因位点变化; 降低序列选择偏好性 | 实验成本较高; 扩增效率和通量较 | [23] |
| 退变寡核苷酸引物PCR | 使用含有退变序列的引物, 在低温退火阶段结合目标DNA的多个位点并启动扩增, 引物3'端包含特定序列, 可随机结合DNA, 通过多轮循环完成DNA扩增 | 降低区域偏好性; 适用于染色体CNV检测和基因克隆 | 随机引物设计可能造成效率低于特异性引物; 基因组覆盖率较低; 对低起始量DNA扩增偏差大 | [24] |
| 多重置换扩增 | 采用Phi29 DNA聚合酶和随机的六聚体引物在恒温条件下扩增DNA, 生成50-100 kb长片段 | 覆盖度和均匀性较好, 不同位点扩增偏差小于3倍; 适用于长片段DNA的高保真扩增, 操作简单 | Phi29 DNA聚合酶昂贵且扩增时间长; 指数扩增可能导致偏倚、覆盖不均 | [25] |
| 转座子介导线性扩增 | 整合Tn5转座酶和T7RNA聚合酶实现单细胞基因组线性扩增. Tn5转座酶随机片段化DNA, T7启动子介导线性扩增, 通过逆转录和第二条链合成双链LIANT产物用于测序 | 扩增均匀性高; 减少扩增偏差; CNV检测精度高; 揭示细胞间变异 | 操作复杂; 存在C-T型假阳性突变 | [26] |
| 多重退火环状循环扩增 | 链置换活性DNA聚合酶生成半扩增子, 随机引物包含27个核苷酸序列和8个可变核苷酸的随机引物, 扩增子头尾互补成环状结构, 58 ℃退火进行扩增, 成环后采用PCR扩增 | 基因组覆盖率高(>95%)且覆盖深度≥1x; 敏感检测CNV和SNV | 需严格控制温度和扩增步骤; 对实验操作环境要求高; 假阳性率高 | [27] |
| 方法 | 技术简介 | 优点 | 缺点 | 期刊 | 参考文献 |
| Tang RNA-seq | 以富含T的多聚核苷酸作为引物合成cDNA, 在3'末端添加Poly(A), 再用poly(T)引物合成第二条cDNA链并扩增, 获得DNA片段 | 检测转录本全长; 高灵敏度和特异性 | 3 '端偏倚, 仅检测带有poly(A)尾的mRNA; 成本高 | Nature methods | [21] |
| Smart-Seq | 低渗溶液裂解单细胞, 将RNA的poly(A)尾与含有Oligo(dT)序列的核苷酸引物杂交, 合成cDNA的第一条链, 全长转录本上添加poly(C), 完成第二条链 | 高覆盖率; 高敏感度, 可识别低、高丰度转录本; 适用稀有细胞 | 低丰度转录本捕获率有限; 不能区分PCR引入的突变, 大于4 kb序列不能高效转录 | Nature methods | [29] |
| Smart-Seq2 | 将oligo(dT)引物与RNA杂交逆转录成cDNA并在3'端添加C尾.利用Tn5转座酶将cDNA片段化, 链接3'端和5'端引物合成cDNA第二条链 | 高覆盖率; 无需纯化; 测序深度为106 | 操作复杂; 对样本质量和完整性要求高; 价格较高 | Nature protocols | [30] |
| CEL-seq | 从单细胞逆转录开始, 利用带有lllumina PI接头、T7启动子、细胞条形码、锚定polyT的引物将mRNA逆转录成cDNA, 第二条链合成后启动IVT构建DNA文库 | 条形码标记可实现高通量分析, 减少扩增偏差; 误差低; 污染小; 灵敏度高、准确、可重复 | 3'端偏倚, 高丰度转录本优先扩增; 操作较复杂 | Nature protocols | [31] |
| Quartz-seq | 将带有Oligo(Dt)引物与mRNA的poly(A)尾杂交, 在反转录酶的作用下合成cDNA的第一条链, cDNA形成发夹结构 | 可定量检测细胞异质性及细胞周期阶段; 高重复性; 减少PCR副产物 | 样本质量要求高和需完整的mRNA; GC含量、长度差异产生扩增偏倚 | Genome biology | [32] |
| MARS-seq | 利用流式细胞术分选单细胞, 随后进行自动化分析, 利用三重条形码标记技术做RNA测序 | 高通量(同时处理数千单细胞); 细胞分类精确 | 技术复杂; mRNA易降解; 扩增偏差 | Science | [33] |
| Drop-seq | 基于微流控的方法将组织解离成单细胞悬液, 将单细胞与STAMP条形码微粒包裹在液滴中. 细胞在液滴中裂解, 释放mRNA携带条形码和不同UMI, 进而逆转录、扩增和测序 | 高吞吐量; 低成本; 通过条形码可追溯细胞来源; 应用广泛 | 细胞丢失和双细胞现象; 有GC含量偏倚 | Nature methods | [34] |
单一组学难以全面解析癌症的异质性本质, 需要单细胞多组学联合应用, 以系统解析癌症的发生发展, 增强研究的可靠性与精确度.
传统批量转录组测序在宏观层面对大量细胞群体进行平均化分析, 从而获得细胞群体的整体基因表达特征. 然而, 该方法存在明显局限性, 它会掩盖低丰度的关键信息, 进而忽略单细胞层面特有的基因表达模式及细胞异质性. 相比之下, SCS技术在解析细胞异质性、追溯细胞发育轨迹以及识别新型细胞亚群等方面具有显著优势, 并已在揭示CRC发生发展机制方面取得重要进展[36]. 尽管如此, SCS技术仍存在一定局限性. 首先, 在技术层面存在数据偏倚风险. 目前SCS的细胞捕获效率仍有待提高, 如10X Genomics Chromium平台的捕获率约为65%, 而BD Rhapsody捕获率仅约为30%[37]. 此外, 在CRC组织解离过程中, 细胞活性易受损, 这可能进一步加剧数据偏倚, 影响结果的可靠性. 因此, 发展更高效率、更高保真性的细胞捕获技术, 仍是未来重要的研究方向.
SCS技术在CRC临床应用方面也面临诸多挑战. 尽管基于多重探针杂交的方法已将检验灵敏度提升至单分子水平, 但其高昂的成本、较长的实验周期及复杂的实验操作流程, 限制了该技术在临床大规模应用推广的可能性[38]. SCS数据本身具有高稀释性和高噪音的特点, 对数据分析提出了更高的要求. 其中之一是批次效应, 即因不同测序平台、文库构建方案、测序运行及试剂批次等引入系统性偏差, 导致不同批次数据之间难以直接比较. 为缓解该问题, 可通过合理设计实验批次分配并结合校正算法, 建立统一的质量控制标准和预处理流程, 从而提高数据的可比性和可靠性. 在CRC研究中, 深入理解肿瘤细胞与TME的相互作用至关重要. 然而, SCS技术无法保留细胞在原始组织中的空间位置信息, 使得研究者难以准确判断不同细胞亚型在组织中的分布及相互间的空间关系. 为攻克这一难题, 将空间转录组学(spatial transcriptomics, ST)技术与SCS技术进行整合, 能够有效定位基因表达的空间分布模式, 有望直观揭示基因相互作用、免疫浸润等关键生物学过程的空间结构基础[39].
肿瘤异质性是指肿瘤细胞增殖过程中因基因组突变、表观遗传修饰及微环境适应性变化, 导致细胞在基因表达、蛋白功能、侵袭能力和治疗响应等方面存在差异[6]. 该特性是驱动肿瘤复发、转移和耐药的关键因素, 也是实现精准治疗的主要障碍[40]. SCS技术对单个肿瘤细胞进行独立高通量分析, 从基因表达谱和表观遗传修饰等层面揭示细胞间差异, 从而深入解析其驱动机制[7]. 该特性不仅影响肿瘤细胞增殖和侵袭能力, 还促进其适应TME的动态变化, 从而推动转移并逃避免疫监视. 深入了解肿瘤异质性对于靶向治疗至关重要.
根据空间分布和来源, 肿瘤异质性可分为两类: 肿瘤内异质性, 指肿瘤内不同细胞间在基因和表型差异, 包括空间和时间两个维度[41]; 以及肿瘤间异质性, 指不同患者或肿瘤类型细胞间的基因与表型差异. 多项研究利用SCS技术揭示了CRC中复杂特征. Dai等[42]通过SCS检测分析鉴定出5个具有独特分子标记的上皮细胞亚群, 证实CRC肿瘤细胞存在高度异质性. SCS技术不仅能揭示样本中核酸多态性的异质性, 还能揭示细胞间的差异, 为肿瘤早发现和早诊断提供了新方法. Liu等[43]比较同一肿瘤不同区域的基因异质性, 探索TME动态变化对肿瘤特征的影响. 发现不同区域在突变谱和拷贝数变异存在差异, 提示空间异质性可能是导致活检诊断结果和药物反应不一致. 这一发现为多点取样或多维度分析在精准诊疗中的应用提供理论依据. Zhao等[44]通过SCS对CRC成纤维细胞进行聚类分析, 鉴定出四种不同分布和功能的亚群, 基于每个亚群的差异表达基因构建了预后风险模型, 为临床风险分层提供参考.
SCS技术不仅是揭示CRC异质性的强大工具, 也为发现靶点、优化诊断策略和预后评估模型构建奠定基础. 但实现临床转化仍需多维度整合与功能验证.
SCS技术在CRC早期诊断和生物标志物中展现出巨大潜力. 相比于传统的液体活检或组织活检, SCS的高分辨率可鉴定罕见细胞亚群和提供更精准的分子图谱, 助力CRC早期筛查. 循环肿瘤细胞(circulating tumor cells, CTCs)是从原发肿瘤或转移灶脱落进入外周血的活跃肿瘤细胞, 具有高度侵袭性和转移潜能. 传统CTCs检测依赖上皮细胞黏附分子等标志物, 可能存在漏检上皮-间质转化(epithelial mesenchymal transition, EMT)的亚群, 而SCS能全面解析CTCs异质性[45], 捕获其转录组和基因组特征. 研究发现, EMT高表达亚群与转移风险显著相关, 提示CTCs可作为液体活检标志物, 实时监测肿瘤进展[46,47]. EMT是上皮细胞向间质细胞转化的关键过程, 伴随细胞顶-基极性丧失、粘附性下降及细胞骨架重塑, 使肿瘤细胞获得侵袭和迁移能力[48]. 该过程受Wnt/β-环连蛋白等经典信号通路和ZEB1、ZEB2等转录因子调控相关, 导致E-钙黏蛋白下调, N-黏钙蛋白和波形蛋白上调, 进一步促进转移. Wei等[49]利用scRNA-seq分析转移性CRC患者的CTCs异质性, 鉴定出高表达EMT基因的亚群, 其表现出更强的侵袭和耐药性. 与此同时, PRL-3也被证实可通过促进EMT增强肿瘤细胞的侵袭和转移能力[50]. 这些结果表明, SCS在解析CTCs异质性和阐明EMT机制方面具有重要价值, 为预测转移风险和制定个体化治疗方案提供了新思路. Smit等[51]通过对CTCs分析, 发现低剂量的Akt抑制剂(MK2206)和mTOR抑制剂(RAD001)能抑制CTCs衍生细胞系生长, 提示PI3K/Akt/mTOR通路是潜在抗CRC转移治疗靶点, 为个体化用药提供了科学依据. 在转移过程中, CTCs还发生代谢重编程, 其代谢特征异质性是驱动转移潜能差异的关键因素[52], 提示对单个CTCs进行代谢组学分析可鉴定转移相关代谢物标志物, 实现基于单细胞代谢特征的CRC转移风险分层.
综上, 通过SCS技术能够提供超越传统单一活检的全面肿瘤信息, 弥补肿瘤分型与分层治疗中的不足. 结合CTCs分析, SCS在早期诊断、预后评估及个体化用药策略中显示出广阔前景, 对推动CRC精准治疗具有重要意义.
TME作为癌细胞赖以生存、发展与演进的动态生态系统, 由免疫细胞、基质细胞、肿瘤细胞、血管和细胞外基质(extracellular matrix, ECM)等共同组成. 这些组分通过细胞间通讯、代谢重编程和免疫调控等机制共同驱动肿瘤进展[5]. 血管网络负责输送氧气与营养物质并清除代谢废物; 基质细胞和免疫细胞分泌多种因子和细胞因子调节肿瘤行为; ECM则提供结构支持并参与信号传导. 通过动态解析TME的细胞异质性及空间互作关系, 能够系统揭示不同肿瘤细胞亚群的分子调控机制及其生物学效应[53].
T细胞作为TME中核心免疫效应细胞, 其功能状态与免疫治疗效果密切相关. 在肿瘤转移早期, 由T/NK细胞介导的免疫反应可抑制肿瘤细胞的迁移; 然而, 在CRC发生肝转移的阶段, T细胞易发生耗竭, 表现为CD4+T细胞数量显著增加而CD8+T细胞数量减少, 从而削弱抗肿瘤免疫应答[54]. Zhang等[55]利用scRNA-seq对CRLM组织及癌旁组织解析, 发现T细胞活化和趋化因子在免疫细胞中亚群中高度活跃, 并鉴定出93个与肿瘤浸润免疫细胞功能失调相关基因. 此外, TME与调控PD-1/PD-L1(programmed cell death protein-1, PD-1/programmed cell death ligand-1, PD-L1)表达水平调控密切相关. 肿瘤相关巨噬细胞(tumor-associated macrophages, TAMs)不仅促进肿瘤细胞表达PD-L1, 肿瘤细胞还可通过调控COX2/mPGES1/PGE2通路诱导TAMs和髓源性抑制性细胞表达PD-L1, 从而增强免疫抑制功能[56]. 综上, TME通过免疫细胞功能失衡及PD-1/PD-L1通路的异常活化, 共同塑造了一个高度免疫抑制环境, 显著影响CRC的进展与免疫治疗反应.
肿瘤相关成纤维细胞(cancer-associated fibroblasts, CAFs)是TME中最丰富的基质细胞, 具有显著功能异质性. 研究表明, CAFs通过分泌多种细胞因子激活PI3K-AKT等信号通路促进EMT, 并通过ECM动态重构与细胞通讯网络促进肿瘤侵袭、转移及耐药. Pelka等[57]应用SCS技术对28名错配修复功能完整型(mismatch repair-proficient, pMMR)患者和34名错配修复功能缺陷型(mismatch repair-deficient, dMMR)CRC患者的癌及癌旁组织测序, 发现两种亚型中均存在炎症表型CAFs亚群, 且在dMMR患者中显著富集, 提示不同错配修复亚型的TME免疫活性在存在差异, 直接影响肿瘤生物学行为及免疫治疗疗效.
CRC的临床进展与治疗反应亦呈现高度异质性. 约50%的CRC患者会发生转移, 且大多数病例无法手术切除. 免疫检查点抑制剂(immune checkpoint inhibitors, ICIs), 如PD-1/PD-L1单抗和抗细胞毒性T淋巴细胞相关抗原-4的单抗, 已在包括CRC在内的多种肿瘤中获批应用[58]. 约15%-20%的患者属于dMMR型, 导致微卫星不稳定(microsatellite instability, MSI). MSI多由错配修复蛋白缺失导致的功能缺陷, 其分为微卫星高度不稳定(microsatellite instability-high, MSI-H)型、微卫星低度不稳定型和微卫星稳定(microsatellite stability, MSS)型. 其中, dMMR/MSI-H因具有较高的肿瘤突变负荷(tumor mutational burden, TMB)和大量免疫细胞浸润而被称之为"热肿瘤", 对ICIs敏感[59]. 帕博利珠单抗单药一线治疗可实现33%的客观缓解率, 为患者带来显著且持久的疗效[60]. 2023年《中国临床肿瘤学会指南》已推荐帕博利珠单抗、纳武利尤单抗联合伊匹木单抗作为dMMR/MSI-H晚期CRC的一线姑息治疗方案, 但是该亚型仅占CRC的5%. 大多数患者为pMMR/MSS型, 其TMB较低和免疫细胞浸润缺乏, 被称为"冷肿瘤", 对单药ICIs应答率低. 因此, 目前临床治疗策略倾向于联合用药. 研究表明, 在一线抗血管内皮生长因子药物联合化疗的基础上加入ICIs, 可显著改善pMMR/MSS型CRC患者的治疗效果[61].
综上, CRC的肿瘤微环境具有高度动态性和复杂的细胞互作网络, 其异质性深刻影响肿瘤进展与免疫应答. 不同MMR亚型的免疫活性差异决定了对免疫检查点抑制剂的敏感性, 也为个体化治疗策略的制定提供了关键依据.
随着SCS技术研究通量的持续提升, 所产生的数据规模呈指数级增长, 其高维、高噪音及高稀疏特征对传统生物信息学分析方法提出了严峻挑战. 在此背景下, 人工智能(artificial intelligence, AI), 特别是机器学习(machine learning, ML)和深度学习(deep learning, DL)正逐渐成为分析和解读单细胞多组学数据不可或缺的工具. 近年来, AI技术在数据预处理、批次效应校正、降维及聚类及细胞类型注释等方面取得显著进展. 在癌症scRNA-seq研究中, ML和DL模型已被应用于区分不同癌症亚型区分、鉴定细胞群体、新的预后生物标志物的发现[62-64]及预测肿瘤抗原免疫原性[65]. 细胞类型注释与功能状态鉴定是解析TME的基础. 传统聚类方法(如PCA、t-SNE/UMAP)高度依赖研究者的先验知识, 处理过程耗时且主观, 对于百万级别细胞大型数据集几乎难以适用. 基于深度学习的自动化算法(如scANVI、CellTypist)通过预训练的模型或迁移算法, 实现对海量细胞快速、精准注释. 相关研究采用CellTypist工具, 成功对近百万个免疫细胞进行了系统注释. 精细刻画了CRC组织中免疫细胞的特异性功能状态. 进一步验证了基于AI与SCS结合策略在识别细胞亚型及实现大规模数据标准化分析方面的巨大潜力, 为深入研究癌症的复杂微环境提供了有力支撑[66].
尽管SCS技术在解析多种癌症的细胞与分子特征等方面取得了重要突破, 但其组织解离过程中会导致细胞空间信息丢失, 仅能获得单一类型的组学信息, 限制了对细胞间的空间相互作用和组织结构的系统解析. ST技术的出现有效弥补了这一不足, 其能够在完整组织切片上构建转录组文库并结合空间定位信息, 使得研究人员在组织切片上定位单个细胞的基因表达谱和空间分布模式[67]. 通过融入空间维度, ST技术极大的增强了对TME结构的精确解析, 提升了临床预后判断的精准性. 这种高分辨率解析涵盖新型细胞与组织特征的识别、基质细胞与免疫细胞的复杂相互作用等, 也为"冷肿瘤"向免疫应答型肿瘤的转化提供了潜在靶点. 多项研究已利用ST技术揭示了CRC相关的新表型特征. 研究发现, CAF-A和CAF-B在CRC中起关键作用, 它们能显著抑制NK细胞功能并破坏细胞因子和趋化因子网络. 在CAFs富集区域, NK细胞数量减少而巨噬细胞显著增多. 进一步机制研究揭示, CAFs通过调节肿瘤相关巨噬细胞间接抑制NK细胞活性[68,69]. Lu等[70]利用公共数据库分析鉴定出转移性CRC中存在特征性癌细胞亚群EP1(KRT17、LAMC2和EMP1高表达)与SPP1+巨噬细胞的交互作用, 诱导PD-L1上调和免疫抑制, 构建促转移微环境. 在另一项ST研究聚焦于肿瘤发生过程中的组织重塑过程, 发现间充质干细胞成纤维细胞亚型减少, 同时FAP+CAF增多并导致代谢异常(如半乳糖、脂肪酸及胆固醇). 与其他研究结论一致, 本研究发现SPP1+巨噬细胞增加与CRC患者较低的无进展生存率相关[71]. 上述研究成果证明了ST技术在深化肿瘤生物学认知中的价值, 为研究肿瘤进展、稳态维持及细胞间相互作用机制提供了关键技术支持. 研究人员正致力于协同利用SCS的高分辨率与ST的空间定位能力, 以系统阐明CRC的空间异质性, 从而最大限度地挖掘数据的潜在价值.
多组合整合策略为更精准的治疗策略奠定了基础. 然而, 这一整合路径面临着挑战: 首先, 在技术层面, 如何将不同分辨率和覆盖度的多数据高效、无偏倚整合; 其次, 在数据分析层面, 海量、多维度对AI算法和数据处理提出更高要求, 需要开发更强大的计算模型; 在临床转化层面, 如何降低技术成本、建立标准化流程并验证其临床效用, 是实现广泛应用前必须跨越的障碍.
SCS技术在CRC的科研探索与临床实践领域意义重大, 其应用体现在: 阐释肿瘤细胞的产生与发展机制, 解析肿瘤及免疫细胞的异质性; 深入剖析动态、复杂的TME, 为免疫学研究提供方法学支持; 并辅助预测患者预后. 与此同时, AI成为从大规模转录组数据中提取真实生物学信息最有力的工具, 在SCS数据分析中推动精准肿瘤学的发展, 指导个体化治疗. 基于当前研究成果, SCS与AI技术的深度整合有望实现CRC的个体化筛查和临床转化. 然而, 该领域仍面临多重挑战. 一方面, AI在数据挖掘过程中面临着信息维度不断扩展所导致需数据量呈指数级增长的问题; 另一方面, SCS技术临床中推广仍受限于高昂的实验成本、单细胞悬液制备复杂、易发细胞污染或损伤等问题. 此外, 由于SCS无法保留细胞空间位置信息, 这一局限性在一定程度上制约了其临床应用深度. 为应对上述挑战, 将SCS与ST技术进行整合, 成为极具前景的发展方向. 这种整合策略将显著增强技术在预后评估、复发风险预测、治疗策略指导和药物开发等临床环节的应用价值. 未来, 亟需开发更经济、高效、自动化的平台和分析技术, 同时建立适用于临床样本质量控制体系. 总之, SCS与AI、ST等前沿技术的多层次整合, 将共同推动CRC的诊疗模式发生根本性变革、从宏观向精准的深刻变革, 并持续引领精准肿瘤学未来的发展方向.
| 1. | Sung H, Ferlay J, Siegel RL, Laversanne M, Soerjomataram I, Jemal A, Bray F. Global Cancer Statistics 2020: GLOBOCAN Estimates of Incidence and Mortality Worldwide for 36 Cancers in 185 Countries. CA Cancer J Clin. 2021;71:209-249. [PubMed] [DOI] |
| 2. | Bray F, Laversanne M, Sung H, Ferlay J, Siegel RL, Soerjomataram I, Jemal A. Global cancer statistics 2022: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA Cancer J Clin. 2024;74:229-263. [PubMed] [DOI] |
| 3. | Siegel RL, Wagle NS, Cercek A, Smith RA, Jemal A. Colorectal cancer statistics, 2023. CA Cancer J Clin. 2023;73:233-254. [PubMed] [DOI] |
| 4. | Zhang A, Zhang Y, Xu J, Zhu R, Liang T, Guo L. Molecular landscape of colorectal cancer liver metastasis: Tumor microenvironment heterogeneity and driver inference. Crit Rev Oncol Hematol. 2025;216:104946. [PubMed] [DOI] |
| 5. | El-Tanani M, Rabbani SA, Babiker R, Rangraze I, Kapre S, Palakurthi SS, Alnuqaydan AM, Aljabali AA, Rizzo M, El-Tanani Y, Tambuwala MM. Unraveling the tumor microenvironment: Insights into cancer metastasis and therapeutic strategies. Cancer Lett. 2024;591:216894. [PubMed] [DOI] |
| 6. | Xu J, Liao K, Yang X, Wu C, Wu W. Using single-cell sequencing technology to detect circulating tumor cells in solid tumors. Mol Cancer. 2021;20:104. [PubMed] [DOI] |
| 7. | Li H, Courtois ET, Sengupta D, Tan Y, Chen KH, Goh JJL, Kong SL, Chua C, Hon LK, Tan WS, Wong M, Choi PJ, Wee LJK, Hillmer AM, Tan IB, Robson P, Prabhakar S. Reference component analysis of single-cell transcriptomes elucidates cellular heterogeneity in human colorectal tumors. Nat Genet. 2017;49:708-718. [PubMed] [DOI] |
| 8. | Valdes-Mora F, Handler K, Law AMK, Salomon R, Oakes SR, Ormandy CJ, Gallego-Ortega D. Single-Cell Transcriptomics in Cancer Immunobiology: The Future of Precision Oncology. Front Immunol. 2018;9:2582. [PubMed] [DOI] |
| 9. | Liu L, Liu C, Quintero A, Wu L, Yuan Y, Wang M, Cheng M, Leng L, Xu L, Dong G, Li R, Liu Y, Wei X, Xu J, Chen X, Lu H, Chen D, Wang Q, Zhou Q, Lin X, Li G, Liu S, Wang Q, Wang H, Fink JL, Gao Z, Liu X, Hou Y, Zhu S, Yang H, Ye Y, Lin G, Chen F, Herrmann C, Eils R, Shang Z, Xu X. Deconvolution of single-cell multi-omics layers reveals regulatory heterogeneity. Nat Commun. 2019;10:470. [PubMed] [DOI] |
| 10. | Guo H, Zhu P, Guo F, Li X, Wu X, Fan X, Wen L, Tang F. Profiling DNA methylome landscapes of mammalian cells with single-cell reduced-representation bisulfite sequencing. Nat Protoc. 2015;10:645-659. [PubMed] [DOI] |
| 11. | Buenrostro JD, Wu B, Litzenburger UM, Ruff D, Gonzales ML, Snyder MP, Chang HY, Greenleaf WJ. Single-cell chromatin accessibility reveals principles of regulatory variation. Nature. 2015;523:486-490. [PubMed] [DOI] |
| 12. | Zhang X, Marjani SL, Hu Z, Weissman SM, Pan X, Wu S. Single-Cell Sequencing for Precise Cancer Research: Progress and Prospects. Cancer Res. 2016;76:1305-1312. [PubMed] [DOI] |
| 13. | Sanger F, Nicklen S, Coulson AR. DNA sequencing with chain-terminating inhibitors. 1977. Biotechnology. 1992;24:104-108. [PubMed] |
| 14. | van Dijk EL, Jaszczyszyn Y, Naquin D, Thermes C. The Third Revolution in Sequencing Technology. Trends Genet. 2018;34:666-681. [PubMed] [DOI] |
| 15. | Tang F, Barbacioru C, Wang Y, Nordman E, Lee C, Xu N, Wang X, Bodeau J, Tuch BB, Siddiqui A, Lao K, Surani MA. mRNA-Seq whole-transcriptome analysis of a single cell. Nat Methods. 2009;6:377-382. [PubMed] [DOI] |
| 16. | Lähnemann D, Köster J, Szczurek E, McCarthy DJ, Hicks SC, Robinson MD, Vallejos CA, Campbell KR, Beerenwinkel N, Mahfouz A, Pinello L, Skums P, Stamatakis A, Attolini CS, Aparicio S, Baaijens J, Balvert M, Barbanson B, Cappuccio A, Corleone G, Dutilh BE, Florescu M, Guryev V, Holmer R, Jahn K, Lobo TJ, Keizer EM, Khatri I, Kielbasa SM, Korbel JO, Kozlov AM, Kuo TH, Lelieveldt BPF, Mandoiu II, Marioni JC, Marschall T, Mölder F, Niknejad A, Rączkowska A, Reinders M, Ridder J, Saliba AE, Somarakis A, Stegle O, Theis FJ, Yang H, Zelikovsky A, McHardy AC, Raphael BJ, Shah SP, Schönhuth A. Eleven grand challenges in single-cell data science. Genome Biol. 2020;21:31. [PubMed] [DOI] |
| 17. | Navin N, Kendall J, Troge J, Andrews P, Rodgers L, McIndoo J, Cook K, Stepansky A, Levy D, Esposito D, Muthuswamy L, Krasnitz A, McCombie WR, Hicks J, Wigler M. Tumour evolution inferred by single-cell sequencing. Nature. 2011;472:90-94. [PubMed] [DOI] |
| 18. | Method of the year 2013. Nat Methods. 2014;11:1. [PubMed] [DOI] |
| 19. | Zhang X, Peng M, Zhu J, Zhai X, Wei C, Jiao H, Wu Z, Huang S, Liu M, Li W, Yang W, Miao K, Xu Q, Chen L, Hu P. Benchmarking metabolic RNA labeling techniques for high-throughput single-cell RNA sequencing. Nat Commun. 2025;16:5952. [PubMed] [DOI] |
| 20. | Zajanckauskaite A, Lingelbach M, Juozapaitė D, Utkus A, Rukšnaitytė G, Jonuškienė G, Gulla A. Utilization of Microfluidic Droplet-Based Methods in Diagnosis and Treatment Methods of Hepatocellular Carcinoma: A Review. Genes (Basel). 2024;15:1242. [PubMed] [DOI] |
| 21. | Tang X, Huang Y, Lei J, Luo H, Zhu X. The single-cell sequencing: new developments and medical applications. Cell Biosci. 2019;9:53. [PubMed] [DOI] |
| 22. | Liu F, Wang Y, Gu H, Wang X. Technologies and applications of single-cell DNA methylation sequencing. Theranostics. 2023;13:2439-2454. [PubMed] [DOI] |
| 23. | Klein CA, Schmidt-Kittler O, Schardt JA, Pantel K, Speicher MR, Riethmüller G. Comparative genomic hybridization, loss of heterozygosity, and DNA sequence analysis of single cells. Proc Natl Acad Sci USA. 1999;96:4494-4499. [PubMed] [DOI] |
| 24. | Telenius H, Carter NP, Bebb CE, Nordenskjöld M, Ponder BA, Tunnacliffe A. Degenerate oligonucleotide-primed PCR: general amplification of target DNA by a single degenerate primer. Genomics. 1992;13:718-725. [PubMed] [DOI] |
| 25. | Dean FB, Hosono S, Fang L, Wu X, Faruqi AF, Bray-Ward P, Sun Z, Zong Q, Du Y, Du J, Driscoll M, Song W, Kingsmore SF, Egholm M, Lasken RS. Comprehensive human genome amplification using multiple displacement amplification. Proc Natl Acad Sci USA. 2002;99:5261-5266. [PubMed] [DOI] |
| 26. | Chen C, Xing D, Tan L, Li H, Zhou G, Huang L, Xie XS. Single-cell whole-genome analyses by Linear Amplification via Transposon Insertion (LIANTI). Science. 2017;356:189-194. [PubMed] [DOI] |
| 27. | Zong C, Lu S, Chapman AR, Xie XS. Genome-wide detection of single-nucleotide and copy-number variations of a single human cell. Science. 2012;338:1622-1626. [PubMed] [DOI] |
| 28. | Nie X, Qin D, Zhou X, Duo H, Hao Y, Li B, Liang G. Clustering ensemble in scRNA-seq data analysis: Methods, applications and challenges. Comput Biol Med. 2023;159:106939. [PubMed] [DOI] |
| 29. | Ramsköld D, Luo S, Wang YC, Li R, Deng Q, Faridani OR, Daniels GA, Khrebtukova I, Loring JF, Laurent LC, Schroth GP, Sandberg R. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 2012;30:777-782. [PubMed] [DOI] |
| 30. | Picelli S, Björklund ÅK, Faridani OR, Sagasser S, Winberg G, Sandberg R. Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nat Methods. 2013;10:1096-1098. [PubMed] [DOI] |
| 31. | Hashimshony T, Senderovich N, Avital G, Klochendler A, de Leeuw Y, Anavy L, Gennert D, Li S, Livak KJ, Rozenblatt-Rosen O, Dor Y, Regev A, Yanai I. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome Biol. 2016;17:77. [PubMed] [DOI] |
| 32. | Sasagawa Y, Nikaido I, Hayashi T, Danno H, Uno KD, Imai T, Ueda HR. Quartz-Seq: a highly reproducible and sensitive single-cell RNA sequencing method, reveals non-genetic gene-expression heterogeneity. Genome Biol. 2013;14:R31. [PubMed] [DOI] |
| 33. | Jaitin DA, Kenigsberg E, Keren-Shaul H, Elefant N, Paul F, Zaretsky I, Mildner A, Cohen N, Jung S, Tanay A, Amit I. Massively parallel single-cell RNA-seq for marker-free decomposition of tissues into cell types. Science. 2014;343:776-779. [PubMed] [DOI] |
| 34. | Islam S, Zeisel A, Joost S, La Manno G, Zajac P, Kasper M, Lönnerberg P, Linnarsson S. Quantitative single-cell RNA-seq with unique molecular identifiers. Nat Methods. 2014;11:163-166. [PubMed] [DOI] |
| 35. | Qian FC, Zhou LW, Zhu YB, Li YY, Yu ZM, Feng CC, Fang QL, Zhao Y, Cai FH, Wang QY, Tang HF, Li CQ. scATAC-Ref: a reference of scATAC-seq with known cell labels in multiple species. Nucleic Acids Res. 2024;52:D285-D292. [PubMed] [DOI] |
| 36. | Yin W, Ao Y, Jia Q, Zhang C, Yuan L, Liu S, Xiao W, Luo G, Shi X, Xin C, Chen M, Lü M, Yu Z. Integrated singlecell and bulk RNA-seq analysis identifies a prognostic signature related to inflammation in colorectal cancer. Sci Rep. 2025;15:874. [PubMed] [DOI] |
| 37. | Salcher S, Heidegger I, Untergasser G, Fotakis G, Scheiber A, Martowicz A, Noureen A, Krogsdam A, Schatz C, Schäfer G, Trajanoski Z, Wolf D, Sopper S, Pircher A. Comparative analysis of 10X Chromium vs. BD Rhapsody whole transcriptome single-cell sequencing technologies in complex human tissues. Heliyon. 2024;10:e28358. [PubMed] [DOI] |
| 38. | Ni Z, Zheng X, Zheng X, Zou X. scLRTD : A Novel Low Rank Tensor Decomposition Method for Imputing Missing Values in Single-Cell Multi-Omics Sequencing Data. IEEE/ACM Trans Comput Biol Bioinform. 2022;19:1144-1153. [PubMed] [DOI] |
| 39. | Wang Y, Qiu X, Li Q, Qin J, Ye L, Zhang X, Huang X, Wen X, Wang Z, He W, Di Y, Zhou Q. Single-cell and spatial-resolved profiling reveals cancer-associated fibroblast heterogeneity in colorectal cancer metabolic subtypes. J Transl Med. 2025;23:175. [PubMed] [DOI] |
| 40. | Aran D. Single-Cell RNA Sequencing for Studying Human Cancers. Annu Rev Biomed Data Sci. 2023;6:1-22. [PubMed] [DOI] |
| 41. | Fisher R, Pusztai L, Swanton C. Cancer heterogeneity: implications for targeted therapeutics. Br J Cancer. 2013;108:479-485. [PubMed] [DOI] |
| 42. | Dai W, Zhou F, Tang D, Lin L, Zou C, Tan W, Dai Y. Single-cell transcriptional profiling reveals the heterogenicity in colorectal cancer. Medicine (Baltimore). 2019;98:e16916. [PubMed] [DOI] |
| 43. | Liu M, Liu Y, Di J, Su Z, Yang H, Jiang B, Wang Z, Zhuang M, Bai F, Su X. Multi-region and single-cell sequencing reveal variable genomic heterogeneity in rectal cancer. BMC Cancer. 2017;17:787. [PubMed] [DOI] |
| 44. | Zhao J, Chen Y. Systematic identification of cancer-associated-fibroblast-derived genes in patients with colorectal cancer based on single-cell sequencing and transcriptomics. Front Immunol. 2022;13:988246. [PubMed] [DOI] |
| 45. | Aceto N, Bardia A, Miyamoto DT, Donaldson MC, Wittner BS, Spencer JA, Yu M, Pely A, Engstrom A, Zhu H, Brannigan BW, Kapur R, Stott SL, Shioda T, Ramaswamy S, Ting DT, Lin CP, Toner M, Haber DA, Maheswaran S. Circulating tumor cell clusters are oligoclonal precursors of breast cancer metastasis. Cell. 2014;158:1110-1122. [PubMed] [DOI] |
| 46. | Gkountela S, Castro-Giner F, Szczerba BM, Vetter M, Landin J, Scherrer R, Krol I, Scheidmann MC, Beisel C, Stirnimann CU, Kurzeder C, Heinzelmann-Schwarz V, Rochlitz C, Weber WP, Aceto N. Circulating Tumor Cell Clustering Shapes DNA Methylation to Enable Metastasis Seeding. Cell. 2019;176:98-112.e14. [PubMed] [DOI] |
| 47. | Hou J, Guo C, Lyu G. Clinical significance of epithelial-mesenchymal transition typing of circulating tumour cells in colorectal cancer. Colorectal Dis. 2020;22:581-587. [PubMed] [DOI] |
| 48. | Xie Y, Wang X, Wang W, Pu N, Liu L. Epithelial-mesenchymal transition orchestrates tumor microenvironment: current perceptions and challenges. J Transl Med. 2025;23:386. [PubMed] [DOI] |
| 49. | Wei C, Yang C, Wang S, Shi D, Zhang C, Lin X, Liu Q, Dou R, Xiong B. Crosstalk between cancer cells and tumor associated macrophages is required for mesenchymal circulating tumor cell-mediated colorectal cancer metastasis. Mol Cancer. 2019;18:64. [PubMed] [DOI] |
| 50. | Lu Y, Chen Y, Wang Z, Shen H, Xu L, Huang C, Tong Y, Shao Y, Zhang H, Fu Z. Single-cell and spatial transcriptome profiling reveal CTHRC1+ fibroblasts promote EMT through WNT5A signaling in colorectal cancer. J Transl Med. 2025;23:282. [PubMed] [DOI] |
| 51. | Smit DJ, Cayrefourcq L, Haider MT, Hinz N, Pantel K, Alix-Panabières C, Jücker M. High Sensitivity of Circulating Tumor Cells Derived from a Colorectal Cancer Patient for Dual Inhibition with AKT and mTOR Inhibitors. Cells. 2020;9:2129. [PubMed] [DOI] |
| 52. | Chen J, Cao S, Situ B, Zhong J, Hu Y, Li S, Huang J, Xu J, Wu S, Lin J, Zhao Q, Cai Z, Zheng L, Wang Q. Metabolic reprogramming-based characterization of circulating tumor cells in prostate cancer. J Exp Clin Cancer Res. 2018;37:127. [PubMed] [DOI] |
| 53. | Hundertmark J, Berger H, Tacke F. Single Cell RNA Sequencing in NASH. Methods Mol Biol. 2022;2455:181-202. [PubMed] [DOI] |
| 54. | Wu K, Zhang G, Shen C, Zhu L, Yu C, Sartorius K, Ding W, Jiang Y, Lu Y. Role of T cells in liver metastasis. Cell Death Dis. 2024;15:341. [PubMed] [DOI] |
| 55. | Zhang Y, Song J, Zhao Z, Yang M, Chen M, Liu C, Ji J, Zhu D. Single-cell transcriptome analysis reveals tumor immune microenvironment heterogenicity and granulocytes enrichment in colorectal cancer liver metastases. Cancer Lett. 2020;470:84-94. [PubMed] [DOI] |
| 56. | Li W, Wu F, Zhao S, Shi P, Wang S, Cui D. Correlation between PD-1/PD-L1 expression and polarization in tumor-associated macrophages: A key player in tumor immunotherapy. Cytokine Growth Factor Rev. 2022;67:49-57. [PubMed] [DOI] |
| 57. | Pelka K, Hofree M, Chen JH, Sarkizova S, Pirl JD, Jorgji V, Bejnood A, Dionne D, Ge WH, Xu KH, Chao SX, Zollinger DR, Lieb DJ, Reeves JW, Fuhrman CA, Hoang ML, Delorey T, Nguyen LT, Waldman J, Klapholz M, Wakiro I, Cohen O, Albers J, Smillie CS, Cuoco MS, Wu J, Su MJ, Yeung J, Vijaykumar B, Magnuson AM, Asinovski N, Moll T, Goder-Reiser MN, Applebaum AS, Brais LK, DelloStritto LK, Denning SL, Phillips ST, Hill EK, Meehan JK, Frederick DT, Sharova T, Kanodia A, Todres EZ, Jané-Valbuena J, Biton M, Izar B, Lambden CD, Clancy TE, Bleday R, Melnitchouk N, Irani J, Kunitake H, Berger DL, Srivastava A, Hornick JL, Ogino S, Rotem A, Vigneau S, Johnson BE, Corcoran RB, Sharpe AH, Kuchroo VK, Ng K, Giannakis M, Nieman LT, Boland GM, Aguirre AJ, Anderson AC, Rozenblatt-Rosen O, Regev A, Hacohen N. Spatially organized multicellular immune hubs in human colorectal cancer. Cell. 2021;184:4734-4752.e20. [PubMed] [DOI] |
| 58. | Johdi NA, Sukor NF. Colorectal Cancer Immunotherapy: Options and Strategies. Front Immunol. 2020;11:1624. [PubMed] [DOI] |
| 59. | Ganesh K, Stadler ZK, Cercek A, Mendelsohn RB, Shia J, Segal NH, Diaz LA. Immunotherapy in colorectal cancer: rationale, challenges and potential. Nat Rev Gastroenterol Hepatol. 2019;16:361-375. [PubMed] [DOI] |
| 60. | Casak SJ, Marcus L, Fashoyin-Aje L, Mushti SL, Cheng J, Shen YL, Pierce WF, Her L, Goldberg KB, Theoret MR, Kluetz PG, Pazdur R, Lemery SJ. FDA Approval Summary: Pembrolizumab for the First-line Treatment of Patients with MSI-H/dMMR Advanced Unresectable or Metastatic Colorectal Carcinoma. Clin Cancer Res. 2021;27:4680-4684. [PubMed] [DOI] |
| 61. | Wu Q, Wang Z, Luo Y, Xie X. Efficacy and safety of immune checkpoint inhibitors in Proficient Mismatch Repair (pMMR)/ Non-Microsatellite Instability-High (non-MSI-H) metastatic colorectal cancer: a study based on 39 cohorts incorporating 1723 patients. BMC Immunol. 2023;24:27. [PubMed] [DOI] |
| 62. | Chen YP, Yin JH, Li WF, Li HJ, Chen DP, Zhang CJ, Lv JW, Wang YQ, Li XM, Li JY, Zhang PP, Li YQ, He QM, Yang XJ, Lei Y, Tang LL, Zhou GQ, Mao YP, Wei C, Xiong KX, Zhang HB, Zhu SD, Hou Y, Sun Y, Dean M, Amit I, Wu K, Kuang DM, Li GB, Liu N, Ma J. Single-cell transcriptomics reveals regulators underlying immune cell diversity and immune subtypes associated with prognosis in nasopharyngeal carcinoma. Cell Res. 2020;30:1024-1042. [PubMed] [DOI] |
| 63. | Zhou Y, Yang D, Yang Q, Lv X, Huang W, Zhou Z, Wang Y, Zhang Z, Yuan T, Ding X, Tang L, Zhang J, Yin J, Huang Y, Yu W, Wang Y, Zhou C, Su Y, He A, Sun Y, Shen Z, Qian B, Meng W, Fei J, Yao Y, Pan X, Chen P, Hu H. Single-cell RNA landscape of intratumoral heterogeneity and immunosuppressive microenvironment in advanced osteosarcoma. Nat Commun. 2020;11:6322. [PubMed] [DOI] |
| 64. | Bao X, Shi R, Zhao T, Wang Y, Anastasov N, Rosemann M, Fang W. Integrated analysis of single-cell RNA-seq and bulk RNA-seq unravels tumour heterogeneity plus M2-like tumour-associated macrophage infiltration and aggressiveness in TNBC. Cancer Immunol Immunother. 2021;70:189-202. [PubMed] [DOI] |
| 65. | Zhou C, Zhu C, Liu Q. Toward in silico Identification of Tumor Neoantigens in Immunotherapy. Trends Mol Med. 2019;25:980-992. [PubMed] [DOI] |
| 66. | Domínguez Conde C, Xu C, Jarvis LB, Rainbow DB, Wells SB, Gomes T, Howlett SK, Suchanek O, Polanski K, King HW, Mamanova L, Huang N, Szabo PA, Richardson L, Bolt L, Fasouli ES, Mahbubani KT, Prete M, Tuck L, Richoz N, Tuong ZK, Campos L, Mousa HS, Needham EJ, Pritchard S, Li T, Elmentaite R, Park J, Rahmani E, Chen D, Menon DK, Bayraktar OA, James LK, Meyer KB, Yosef N, Clatworthy MR, Sims PA, Farber DL, Saeb-Parsy K, Jones JL, Teichmann SA. Cross-tissue immune cell analysis reveals tissue-specific features in humans. Science. 2022;376:eabl5197. [PubMed] [DOI] |
| 67. | Saviano A, Henderson NC, Baumert TF. Single-cell genomics and spatial transcriptomics: Discovery of novel cell states and cellular interactions in liver physiology and disease biology. J Hepatol. 2020;73:1219-1230. [PubMed] [DOI] |
| 68. | Peng Z, Ye M, Ding H, Feng Z, Hu K. Spatial transcriptomics atlas reveals the crosstalk between cancer-associated fibroblasts and tumor microenvironment components in colorectal cancer. J Transl Med. 2022;20:302. [PubMed] [DOI] |
| 69. | Zhang R, Qi F, Zhao F, Li G, Shao S, Zhang X, Yuan L, Feng Y. Cancer-associated fibroblasts enhance tumor-associated macrophages enrichment and suppress NK cells function in colorectal cancer. Cell Death Dis. 2019;10:273. [PubMed] [DOI] |
| 70. | Fu H, Lu X, Ji T, Wang L, Wang G, Wang L, Wang Z. Integrated analysis of colorectal cancer metastasis identifies characteristics of tumor cell during metastasis. Gastroenterol Rep (Oxf). 2024;12:goae055. [PubMed] [DOI] |
| 71. | Qi J, Sun H, Zhang Y, Wang Z, Xun Z, Li Z, Ding X, Bao R, Hong L, Jia W, Fang F, Liu H, Chen L, Zhong J, Zou D, Liu L, Han L, Ginhoux F, Liu Y, Ye Y, Su B. Single-cell and spatial analysis reveal interaction of FAP(+) fibroblasts and SPP1(+) macrophages in colorectal cancer. Nat Commun. 2022;13:1742. [PubMed] [DOI] |
学科分类: 胃肠病学和肝病学
手稿来源地: 河北省
同行评议报告学术质量分类
A级 (优秀): 0
B级 (非常好): B, B, B, B
C级 (良好): C, C, C, C
D级 (一般): 0
E级 (差): E
科学编辑: 刘继红 制作编辑:张砚梁