修回日期: 2025-04-21
接受日期: 2025-05-08
在线出版日期: 2025-05-28
机器学习(machine learning, ML)通过强大的数据处理能力在预测各种临床事件中取得良好表现. 本研究旨在利用临床和超声内镜数据开发ML模型以实现胰腺良恶性病变的准确预测, 并通过沙普利加和解释(SHapley Additive exPlanations, SHAP)方法解释ML模型.
根据超声内镜影像组学特征和临床特征开发可解释的机器学习模型, 以预测胰腺病变的良恶性.
收集天津医科大学总医院2014-01/2024-12接受超声内镜检查的共216名胰腺病变患者的超声内镜图像及临床信息, 其中训练集150名, 验证集66名. 应用t检验和最小绝对收缩和选择运算符逻辑回归算法选择超声内镜影像组学特征并构建5种影像组学机器学习模型, 选择性能最优的极端梯度提升(extreme gradient boosting, XGBoost)模型以进行后续分析. 采用单因素和多因素Logistic回归分析训练集胰腺良恶性病变中有统计学差异的临床指标以构建临床XGBoost模型. 最后, 联合影像组学和临床特征构建多模态联合XGBoost模型. 采用SHAP工具探索模型的可解释性.
影像组学XGBoost机器学习模型由15个影像组学特征构建, 训练集和验证集曲线下面积(area under curve, AUC)分别为0.8521和0.8761. 临床XGBoost机器学习模型由3个临床特征构成, 训练集和验证集的AUC分别为0.9286和0.9200. 多模态机器学习模型由上述15个影像组学特征和3个临床特征构成, 其在训练集和验证集的AUC分别为0.9458和0.9357, 优于上述2种模型. SHAP分析表明多模态机器学习模型中贡献度居前5位的特征或指标包括3项临床实验室指标和2项影像组学特征.
联合影像组学和临床特征的多模态机器学习模型, 可以有效预测胰腺病变的良恶性, SHAP工具将预测过程可视化以供临床应用.
核心提要: 我们收集了天津医科大学总医院2014-01/2024-12接受超声内镜检查的共216名胰腺病变患者的超声内镜图像及临床信息, 基于超声内镜影像组学特征和临床特征, 构建了多模态机器学习模型以识别胰腺病变的良恶性, 并用沙普利加和解释分析以探索模型的可解释性.
引文著录: 蔡晓晗, 范晓飞, 李姝, 方维丽, 王邦茂, 王玉峰, 冯月, 穆金宝, 刘文天. 基于影像组学和临床特征构建用于区分胰腺良恶性病变的多模态可解释机器学习模型. 世界华人消化杂志 2025; 33(5): 361-372
Revised: April 21, 2025
Accepted: May 8, 2025
Published online: May 28, 2025
Machine learning (ML) has achieved good performance in predicting various clinical events due to its powerful data processing capabilities. This study aimed to develop ML models using clinical and endoscopic ultrasound data to accurately predict benign and malignant pancreatic lesions, and to interpret these models by applying the SHapley Additive exPlanations (SHAP) method.
To develop an interpretable machine learning model based on endoscopic ultrasonography (EUS) radiomics features and clinical features to predict the benign and malignant nature of pancreatic lesions.
We collected EUS images and clinical information from 216 patients with pancreatic lesions who underwent EUS examination at Tianjin Medical University General Hospital from January 2014 to December 2024, including a training set of 150 patients and a validation set of 66 patients. We used t-tests and the least absolute shrinkage and selection operator logistic regression algorithm to select EUS radiomics features and constructed five radiomics-based machine learning models, ultimately selecting the extreme gradient boosting (XGBoost) model with the best performance for further analysis. Univariate and multivariate logistic regression analyses were used to identify statistically significant clinical indicators distinguishing benign and malignant pancreatic lesions in the training set, and a clinical feature-based XGBoost model was developed. Finally, a multimodal combined XGBoost model was constructed by integrating radiomics and clinical features. The SHAP method was used to explore the interpretability of the model.
The radiomics-based XGBoost machine learning model was constructed with 15 radiomics features, achieving area under the curve (AUC) values of 0.8521 and 0.8761 for the training and validation sets, respectively. The clinical feature-based XGBoost machine learning model consisted of three clinical features, with AUC values of 0.9286 and 0.9200 for the training and validation sets, respectively. The multimodal machine learning model included the aforementioned 15 radiomics features and three clinical features, yielding AUC values of 0.9458 and 0.9357 for the training and validation sets, outperforming the other two models. SHAP analysis indicated that the top five contributing features or indicators in the machine learning model included three clinical laboratory indicators and two radiomics features.
The multimodal machine learning model that combines EUS radiomics and clinical features can effectively predict the benign and malignant nature of pancreatic lesions, and the SHAP tool visualizes the prediction process for clinical application.
- Citation: Cai XH, Fan XF, Li S, Fang WL, Wang BM, Wang YF, Feng Y, Mu JB, Liu WT. Construction of a multimodal interpretable machine learning model based on radiomics and clinical features for distinguishing benign and malignant pancreatic lesions. Shijie Huaren Xiaohua Zazhi 2025; 33(5): 361-372
- URL: https://www.wjgnet.com/1009-3079/full/v33/i5/361.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v33.i5.361
胰腺癌和胰腺良性病变如慢性胰腺炎、自身免疫性胰腺炎等是常见的胰腺病变[1]. 其中胰腺癌在2022年全球癌症统计中占癌症相关死亡率第6名[2], 5年生存率低至10%[3], 具有高度恶性、进展迅速、预后差的特征[4], 手术切除可能是根治胰腺癌的唯一方法[5]. 而胰腺良性病变预后相对较好, 不恰当的手术切除可能导致胰腺良性病变患者出现不必要的风险和并发症[6]. 因此准确区分胰腺病变的良恶性对胰腺的治疗及预后非常重要.
超声内镜(endoscopic ultrasonography, EUS)是诊断胰腺病变的一种有价值技术, 与电脑扫描(computed tomography, CT)或磁共振成像(magnetic resonance imaging, MRI)相比, 其可呈现高分辨率和更详细的胰腺空间成像, 为小胰腺肿块和囊肿的成像提供了额外优势[7]. 但其诊断性能很大程度上受医生水平影响, 观察者内及观察者间的差异也使影像的解读受到挑战[8]. EUS引导下的细针穿刺或活检显著提高了整体诊断准确性, 但因其为侵入性检查, 可引起出血、感染、自限性胰腺炎等并发症[9]. 因此需要一些其他技术为临床胰腺病变的诊断提供更多帮助.
机器学习(machine learning, ML)是人工智能的一个子集, 其是一种计算机程序, 使机器能够在没有显式编程的情况下进行学习[10]. 与传统的统计方法相比, 其可处理复杂的非线性和交互关系, 从而对临床信息提供深入见解[11]. 其在临床预测方面已取得良好表现[12-14], 但上述模型仅在单一模式上运行, 忽略了其他指标(如实验室化验结果)对诊断的潜在影响. 同时传统的机器学习往往由于缺乏可解释性导致"黑匣子"问题, 不利于临床应用[15,16]. 因此本研究基于胰腺超声内镜影像组学和临床实验室指标, 构建鉴别胰腺良恶性病变的多模态机器学习诊断模型, 并用沙普利加和解释(SHapley Additive exPlanations, SHAP)法实现模型的可解释性.
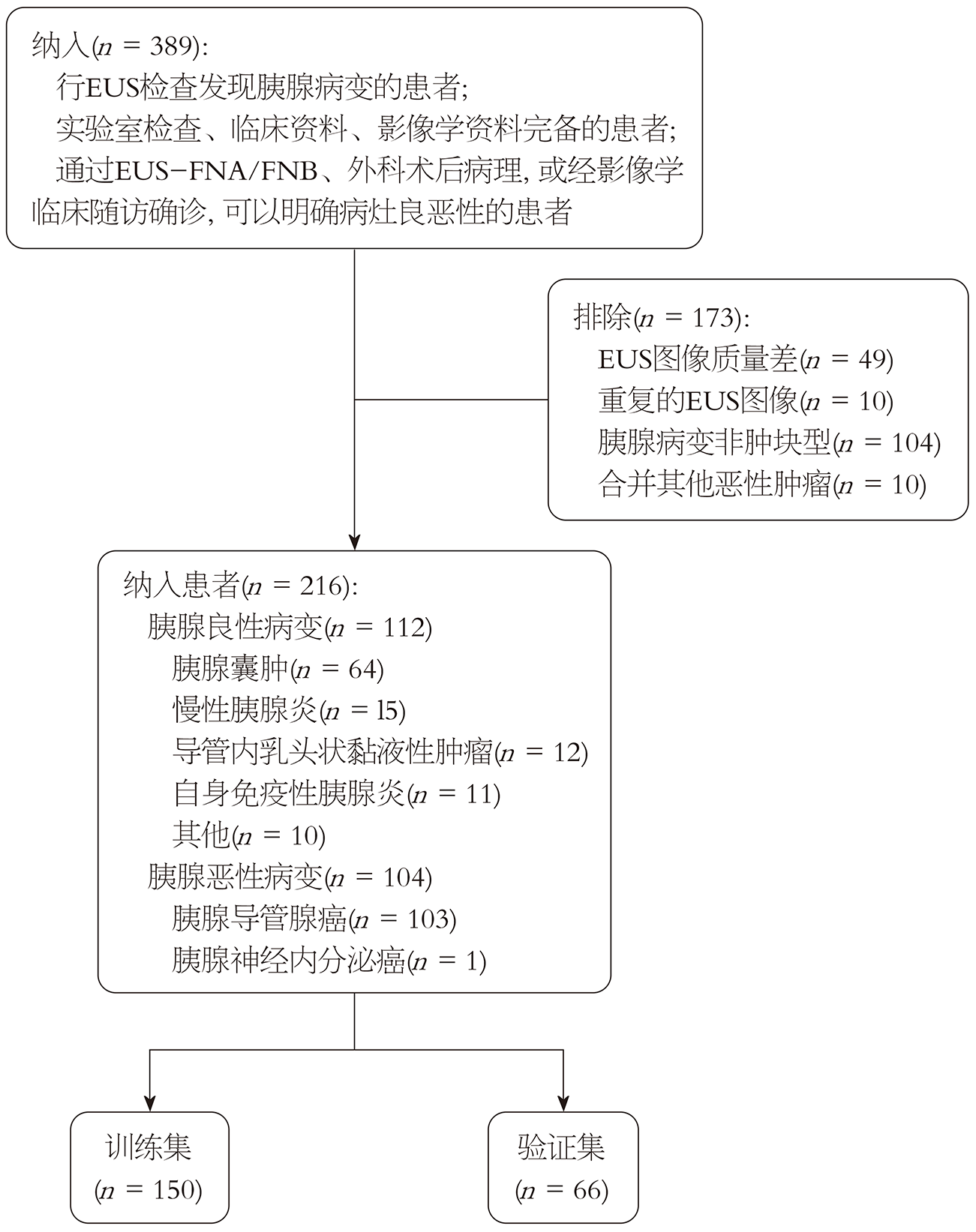
1.1.1 研究对象: 本研究回顾性收集了天津医科大学总医院2014-01/2024-12共389例胰腺病变患者的临床和超声内镜图像信息, 其中216名患者被纳入最终分析. 该研究的纳入标准如下: (1)行EUS检查发现胰腺病变的患者; (2)患者的实验室检查、临床资料、影像学资料完备; (3)通过EUS-FNA/FNB、外科术后病理, 或经影像学临床随访确诊, 可以明确病灶良恶性的患者. 排除标准: (1)EUS图像质量差, 不能用于分析; (2)重复的EUS检查图像; (3)非肿块型胰腺病变的患者. 具体流程见图1(图中其他包括4例胰腺神经内分泌瘤, 3例浆液性囊腺瘤, 3例黏液性囊性肿瘤). 本研究已获得天津医科大学总医院医院伦理委员会的批准(参考编号: IRB2022-YX-249-01).
1.1.2 临床特征的收集和筛选: 从本院病历档案中收集记录了每位患者的以下临床资料: 年龄(岁)、病变大小(厘米)、糖类抗原199(carbohydrate antigen 199, CA199)(U/mL)、直接胆红素(μmol/L)、癌胚抗原(ng/mL)、血糖(mmol/L)、钙(mmol/L)、白细胞(×109/L)、红细胞(×109/L)、血红蛋白(g/L)、血小板(×109/L)、总蛋白(g/L)、白蛋白(g/L)、球蛋白(g/L)、谷丙转氨酶(U/L)、谷草转氨酶(U/L)、碱性磷酸酶(U/L)、性别(男、女)、糖化血红蛋白(正常、升高)、病变部位(胰腺头颈部、胰腺体尾部、全部胰腺). 采用单因素和多因素Logistic分析确定与胰腺良恶性相关的重要临床因素.
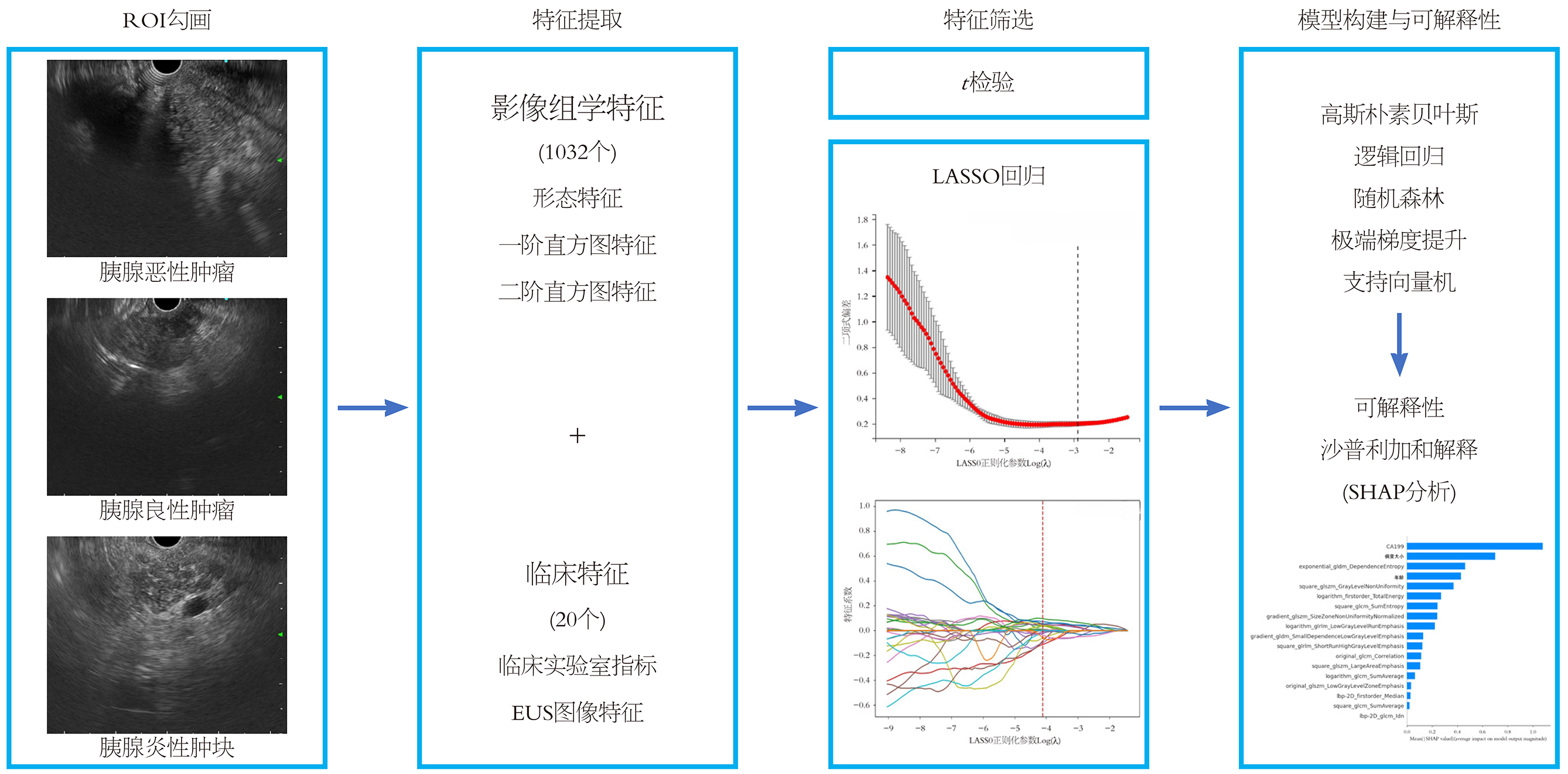
1.1.3 EUS图像采集与分割: 所有患者接受环扫EUS(GF-UCT240-AL5; Olympus Medical, Tokyo, Japan)或扇扫EUS(GF-UE260-AL5; Olympus Medical, Tokyo, Japan)检查. 术前排除EUS及穿刺的禁忌证, 向患者及家属充分告知操作相关风险, 并签署知情同意书. 由高年资的EUS医师以盲法方式对胰腺进行连续性扫查, 探查了病变部位(胰腺头颈、体尾、全部)、病变大小(最大径)等特征, 将采集到的EUS图像导入COCO Annotator软件中手动勾画感兴趣区(regions of Interest, ROI), 并对上述图像进行图像灰度化处理.
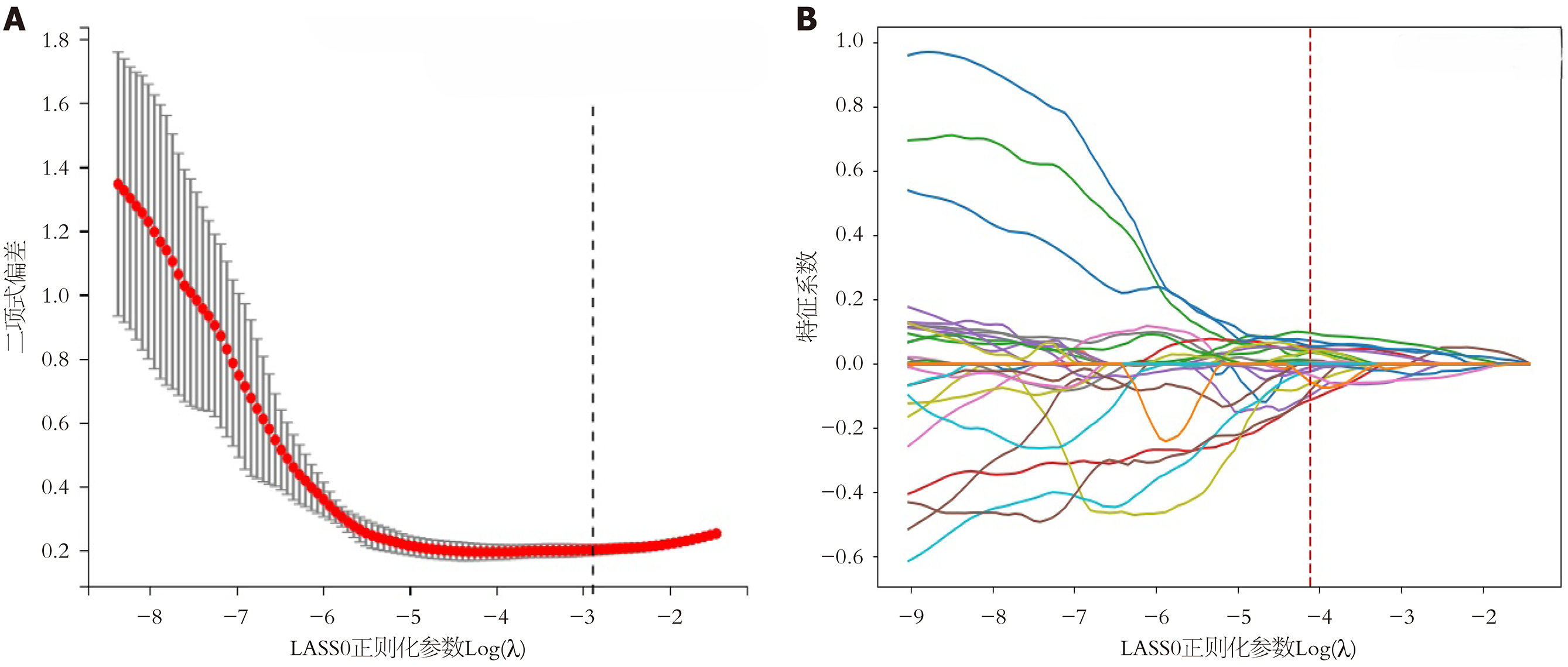
1.2.1 影像组学特征提取与筛选: 使用 Python 3.8.0和Pyradiomic自动提取经灰度化处理的ROI中的影像组学特征, 以描述病灶的形状、性质、位置和与周围组织的关系等. 本研究中每张EUS图像的ROI均提取到1032个影像组学特征, 可依据特征类型分为三类: (1)形态特征; (2)一阶直方图特征; (3)二阶直方图特征, 包括灰度共生矩阵, 灰度依赖矩阵, 灰度游程矩阵, 灰度级大小区域矩阵和邻域灰度差矩阵. 采取了两个连续的步骤, 将放射组学特征的维数降低到一个可管理的数量. 首先, 通过t检验确定了胰腺良恶性病变之间具有统计学意义的放射组学特征. 随后, 为了最大限度地减少冗余, 采用最小绝对收缩和选择运算符逻辑(Least Absolute Shrinkage and Selection Operator, LASSO)回归算法进一步进行特征筛选, 并执行5倍交叉验证选择最优参数以获得最优影像组学模型.
1.2.2 模型构建: 利用上述筛选出的影像组学特征, 在训练集中开发了5种影像组学机器学习模型: 高斯朴素贝叶斯(gaussian naive bayes, GNB)、逻辑回归(logistic regression, LR)、随机森林(random forest, RF)、支持向量机(support vector machine, SVM)和极端梯度提升(extreme gradient boosting, XGBoost). 使用5倍交叉验证确定模型中的最佳参数. 模型的主要评估指标包括接收者操作特征曲线(rceiver operating characteristic, ROC)曲线下面积(area under curve, AUC)、准确性、敏感性、特异性、F1. 根据上述结果, 选择了最具预测性的机器学习分类器. 此外, 收集有意义的临床信息和EUS病变特征构建了一个临床预测模型; 最后将影像组学特征与临床信息相结合, 建立多模态联合预测模型. 通过SHAP分析计算每个特征对联合机器学习模型输出的影响以实现对模型的可解释性. 具体工作流程如图2所示.
统计学处理 本研究采用SPSS 26.0及Python进行统计学分析. 符合正态分布及近似正态分布的计量资料用均值±标准差描述, 不符合正态分布的计量资料则表示为中位数(四分位数间距); 计数资料用例数(百分比)表示. 使用Student's t检验或Mann-Whitney U检验对连续变量进行评估, 使用χ2检验或Fisher精确检验对分类变量进行评估. 多因素回归分析确定了具有统计学差异的临床特征, 用于开发临床预测模型. 用AUC、准确性、敏感性、特异性、阳性预测值和阴性预测值评估每个模型的性能. P<0.05具有统计学差异.
2014-01至2024-12从我院共收集了439名胰腺病变患者, 在应用上述排除与纳入标准后, 共有216名患者被纳入(111名男性, 105名女性). 其中训练集有150人(69.4%), 验证集有66例(30.6%). 训练集中胰腺良恶性病变患者的年龄、病变大小、CA199、直接胆红素、癌胚抗原、血糖、白细胞、碱性磷酸酶具有统计学差异(P<0.05). 而2组血钙、红细胞、血红蛋白、血小板、总蛋白、白蛋白、球蛋白、谷丙转氨酶、谷草转氨酶、性别、糖化血红蛋白、病变部位无统计学差异(P>0.05). 表1详细说明了训练集和验证集中患者的特征.
| 变量 | 训练集(n = 150) | 验证集(n = 66) | ||||||
| 良性(n = 78) | 恶性(n = 72) | 统计量 | P值 | 良性(n = 34) | 恶性(n = 32) | 统计量 | P值 | |
| 年龄[岁, M(Q1, Q3)] | 60.00(46.00, 64.00) | 63.50(55.50, 67.00) | Z = -3.17 | 0.002a | 66.50(58.00, 68.00) | 68.00(58.00, 70.00) | Z = -1.43 | 0.152 |
| 病变大小[厘米, M(Q1, Q3)] | 2.40(1.33, 3.70) | 3.85(3.10, 4.65) | Z = -5.10 | <0.001a | 2.40(1.21, 3.24) | 3.60(3.08, 4.82) | Z = -3.87 | <0.001a |
| CA199[U/mL, M(Q1, Q3)] | 21.15(7.36, 157.18) | 467.67(67.67, 1200.00) | Z = -5.86 | <0.001a | 8.90(3.78, 21.15) | 855.15(185.05, 1200.00) | Z = -6.07 | <0.001a |
| 直接胆红素[μmol/L, M(Q1, Q3)] | 2.90(2.90, 4.70) | 4.50(2.95, 12.20) | Z = -2.56 | 0.011a | 2.95(2.62, 4.52) | 4.30(2.68, 7.25) | Z = -1.70 | 0.090 |
| 癌胚抗原[ng/mL, M(Q1, Q3)] | 2.02(1.89, 3.10) | 4.69(2.93, 9.27) | Z = -5.84 | <0.001a | 1.94(1.77, 2.26) | 5.85(3.82, 6.55) | Z = -4.92 | <0.001a |
| 血糖[mmol/L, M(Q1, Q3)] | 4.95(4.95, 6.00) | 5.80(5.15, 6.62) | Z = -3.11 | 0.002a | 6.30(5.50, 6.30) | 5.40(5.38, 6.35) | Z = -1.39 | 0.163 |
| 钙[mmol/L, M(Q1, Q3)] | 2.25(2.23, 2.26) | 2.27(2.20, 2.33) | Z = -0.99 | 0.321 | 2.35(2.21, 2.35) | 2.31(2.26, 2.42) | Z = -0.56 | 0.575 |
| 白细胞[×109/L, M(Q1, Q3)] | 5.48(5.15, 5.58) | 6.43(4.39, 7.58) | Z = -2.26 | 0.024a | 6.07(5.11, 6.10) | 6.58(5.75, 7.70) | Z = -2.41 | 0.016a |
| 红细胞[×109/L, M(Q1, Q3)] | 4.27(4.11, 4.34) | 4.15(3.71, 4.57) | Z = -0.66 | 0.508 | 4.08(3.84, 4.08) | 4.22(3.88, 4.70) | Z = -1.89 | 0.059 |
| 血红蛋白[g/L, M(Q1, Q3)] | 127.00(122.00, 131.00) | 124.50(112.00, 139.00) | Z = -0.51 | 0.607 | 125.00(119.75, 125.00) | 125.50(117.25, 138.00) | Z = -0.98 | 0.327 |
| 血小板[×109/L, M(Q1, Q3)] | 197.00(186.75, 210.00) | 200.00(147.75, 261.50) | Z = -0.75 | 0.456 | 239.00(227.25, 243.00) | 229.50(187.50, 279.00) | Z =-0.77 | 0.440 |
| 总蛋白[g/L, M(Q1, Q3)] | 68.00(64.00, 68.00) | 68.00(62.00, 72.00) | Z = -0.86 | 0.389 | 64.00(63.25, 68.75) | 68.50(64.75, 74.00) | Z = -1.84 | 0.066 |
| 白蛋白[g/L, M(Q1, Q3)] | 40.00(36.00, 40.00) | 38.50(36.00, 42.00) | Z = -0.68 | 0.497 | 38.00(35.00, 38.75) | 38.00(36.00, 41.00) | Z = -1.23 | 0.217 |
| 球蛋白[g/L, M(Q1, Q3)] | 31.00(27.00, 31.00) | 28.50(25.00, 32.00) | Z = -0.78 | 0.436 | 28.00(27.00, 31.75) | 29.00(26.00, 32.25) | Z = -0.84 | 0.402 |
| 谷丙转氨酶[U/L, M(Q1, Q3)] | 15.00(15.00, 33.25) | 23.00(13.75, 61.25) | Z = -1.82 | 0.068 | 14.00(12.25, 17.75) | 16.50(11.75, 44.25) | Z = -1.40 | 0.162 |
| 谷草转氨酶[U/L, M(Q1, Q3)] | 20.00(17.00, 20.75) | 19.00(15.00, 64.50) | Z = -0.58 | 0.560 | 17.00(16.25, 19.75) | 21.50(15.75, 44.75) | Z = -1.53 | 0.127 |
| 碱性磷酸酶[U/L, M(Q1, Q3)] | 74.00(74.00, 88.75) | 107.00(73.00, 195.00) | Z = -3.64 | <0.001a | 76.00(72.25, 93.50) | 97.00(77.00, 246.75) | Z = -2.56 | 0.011a |
| 糖化血红蛋白[例(%)] | χ2 = 0.07 | 0.791 | χ2 = 0.30 | 0.585 | ||||
| 正常 | 61(78.21) | 55(76.39) | 19(55.88) | 20(62.50) | ||||
| 升高 | 17(21.79) | 17(23.61) | 15(44.12) | 12(37.50) | ||||
| 性别[例(%)] | χ2 = 1.36 | 0.244 | χ2 = 0.97 | 0.325 | ||||
| 男 | 37(47.44) | 41(56.94) | 15(44.12) | 18(56.25) | ||||
| 女 | 41(52.56) | 31(43.06) | 19(55.88) | 14(43.75) | ||||
| 病变部位[例(%)] | χ2 = 0.20 | 0.903 | - | 0.101 | ||||
| 胰腺头颈部 | 37(47.44) | 34(47.22) | 16(47.06) | 22(68.75) | ||||
| 胰腺体尾部 | 34(43.59) | 30(41.67) | 15(44.12) | 10(31.25) | ||||
| 全部胰腺 | 7(8.97) | 8(11.11) | 3(8.82) | 0(0.00) | ||||
最初从每张EUS图像ROI中共提取了1032个放射组学特征. 通过t检验筛选出300个有显著差异的特征. 随后使用LASSO回归进一步筛选, 其通过制定包含惩罚项的优化目标函数来完成变量选择和复杂性调整, 最终保留了15个稳健的放射组学特征, 详见图3A和B. 利用最终筛选的15个影像组学特征开发了5种机器学习模型, 包括GNB、LR、RF、XGBoost、SVM. 其中LR、RF和XGBoost模型在训练集中表现较佳, 但在验证集中XGBoost模型的AUC为0.8761、准确性为0.7955、敏感性为0.7206、特异性为0.8750、F1分数为0.7840, 优于LR和RF模型. 综合考虑训练集和验证集性能, XGBoost模型表现更稳定优异(详见表2和表3).
| 机器学习模型 | AUC | 准确性 | 敏感性 | 特异性 | F1分数 |
| GNB | 0.8250 | 0.8333 | 0.7333 | 0.9583 | 0.8302 |
| LR | 0.8819 | 0.7963 | 0.7333 | 0.8750 | 0.8000 |
| RF | 0.8500 | 0.8148 | 0.8000 | 0.8750 | 0.8421 |
| XGBoost | 0.8521 | 0.8333 | 0.7333 | 0.8750 | 0.8000 |
| SVM | 0.8369 | 0.7931 | 0.6667 | 0.9167 | 0.7692 |
| 机器学习模型 | AUC | 准确性 | 敏感性 | 特异性 | F1分数 |
| GNB | 0.7229 | 0.6212 | 0.7794 | 0.4531 | 0.6795 |
| LR | 0.8196 | 0.7727 | 0.6765 | 0.8750 | 0.7541 |
| RF | 0.8095 | 0.7576 | 0.6471 | 0.8750 | 0.7333 |
| XGBoost | 0.8761 | 0.7955 | 0.7206 | 0.8750 | 0.7840 |
| SVM | 0.7096 | 0.6288 | 0.6176 | 0.6406 | 0.6316 |
对训练集患者的临床实验室指标和EUS图像特征进行单因素Logistic回归分析, 选取P<0.05的指标进行多因素Logistic回归分析. 单因素Logistic回归结果表明, 年龄、CA199、病变最大径、谷丙转氨酶、谷草转氨酶和碱性磷酸酶与胰腺病变的良恶性鉴别相关(P<0.05). 多因素回归分析表明年龄(OR = 1.05, 95%CI: 1.01-1.09, P = 0.008)、CA199(OR = 1.27, 95%CI: 1.06-1.54, P = 0.011)、病变最大径(OR = 1.01, 95%CI: 1.01-1.01, P<0.001)是影响胰腺良恶性病变诊断的因素(详见表4). 因此基于上述3个特征构建临床XGBoost机器学习模型, 该模型在训练集和测试集中的AUC分别为0.9286和0.9200.
| 变量variables | 单因素逻辑回归 | 多因素逻辑回归 | ||||||||
| β | S.E | Z | P值 | OR(95%CI) | β | S.E | Z | P值 | OR(95%CI) | |
| 年龄[岁, M(Q1, Q3)] | 0.04 | 0.01 | 3.09 | 0.002a | 1.05(1.02-1.08) | 0.05 | 0.02 | 2.64 | 0.008a | 1.05(1.01-1.09) |
| 病变大小[厘米, M(Q1, Q3)] | 0.28 | 0.10 | 2.85 | 0.004a | 1.33(1.09-1.61) | 0.24 | 0.09 | 2.56 | 0.011a | 1.27(1.06-1.54) |
| CA199 [U/mL, M(Q1, Q3)] | 0.01 | 0.00 | 5.12 | <0.001a | 1.01(1.01-1.01) | 0.01 | 0.00 | 4.63 | <0.001a | 1.01(1.01-1.01) |
| 直接胆红素[μmol/L, M(Q1, Q3)] | 0.01 | 0.00 | 1.58 | 0.114 | 1.01(1.00-1.01) | |||||
| 癌胚抗原[ng/mL, M(Q1, Q3)] | 0.01 | 0.01 | 1.39 | 0.163 | 1.01(1.00-1.02) | |||||
| 血糖[mmol/L, M(Q1, Q3)] | 0.04 | 0.05 | 0.85 | 0.397 | 1.04(0.95-1.15) | |||||
| 钙[mmol/L, M(Q1, Q3)] | -0.16 | 0.25 | -0.64 | 0.520 | 0.85(0.53-1.39) | |||||
| 白细胞[×109/L, M(Q1, Q3)] | 0.15 | 0.09 | 1.67 | 0.095 | 1.16(0.98-1.37) | |||||
| 红细胞[×109/L, M(Q1, Q3)] | -0.39 | 0.32 | -1.20 | 0.230 | 0.68(0.36-1.28) | |||||
| 血红蛋白[g/L, M(Q1, Q3)] | -0.01 | 0.01 | -0.65 | 0.515 | 0.99(0.97-1.01) | |||||
| 血小板[×109/L, M(Q1, Q3)] | 0.00 | 0.00 | 1.70 | 0.088 | 1.00(1.00-1.01) | |||||
| 总蛋白[g/L, M(Q1, Q3)] | 0.02 | 0.03 | 0.82 | 0.411 | 1.02(0.97-1.08) | |||||
| 白蛋白[g/L, M(Q1, Q3)] | 0.01 | 0.04 | 0.16 | 0.875 | 1.01(0.94-1.08) | |||||
| 球蛋白[g/L, M(Q1, Q3)] | -0.03 | 0.04 | -0.79 | 0.430 | 0.97(0.91-1.04) | |||||
| 谷丙转氨酶[U/L, M(Q1, Q3)] | 0.01 | 0.00 | 2.26 | 0.024a | 1.01(1.01-1.01) | 0.00 | 0.01 | 0.87 | 0.385 | 1.00(0.99-1.01) |
| 谷草转氨酶[U/L, M(Q1, Q3)] | 0.01 | 0.00 | 2.12 | 0.034a | 1.01(1.01-1.01) | -0.00 | 0.01 | -0.49 | 0.623 | 1.00(0.98-1.01) |
| 碱性磷酸酶[U/L, M(Q1, Q3)] | 0.01 | 0.00 | 2.48 | 0.013a | 1.01(1.01-1.01) | 0.00 | 0.00 | 1.35 | 0.176 | 1.00(1.00-1.01) |
| 糖化血红蛋白[例(%)] | ||||||||||
| 正常 | 1.00(reference) | |||||||||
| 升高 | 0.10 | 0.39 | 0.27 | 0.791 | 1.11(0.52-2.38) | |||||
| 性别[例(%)] | ||||||||||
| 男 | 1.00(reference) | |||||||||
| 女 | -0.38 | 0.33 | -1.16 | 0.245 | 0.68(0.36-1.30) | |||||
| 病变部位[例(%)] | ||||||||||
| 胰腺头颈部 | 1.00(reference) | |||||||||
| 胰腺体尾部 | -0.04 | 0.35 | -0.12 | 0.906 | 0.96(0.49-1.89) | |||||
| 全部胰腺 | 0.22 | 0.57 | 0.38 | 0.702 | 1.24(0.41-3.80) | |||||
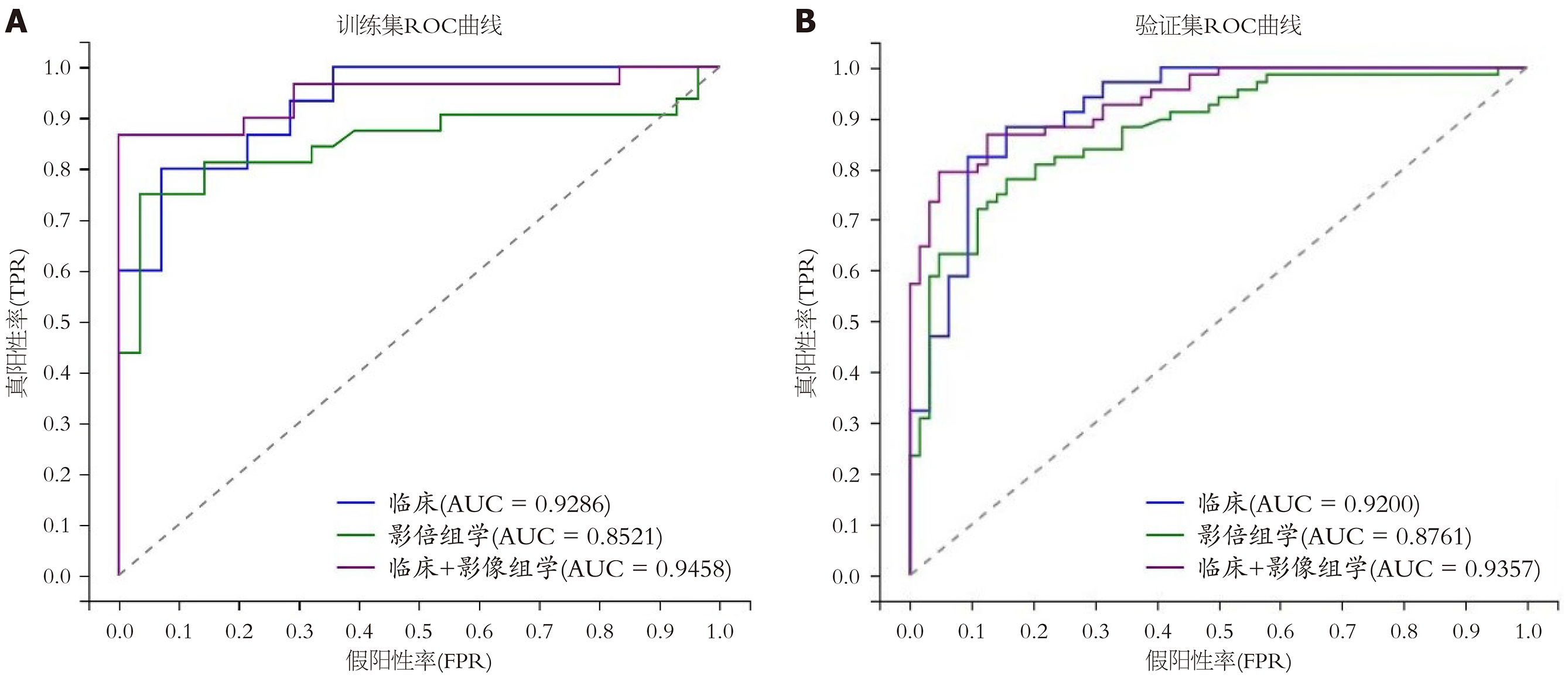
利用XGBoost机器学习模型开发一个包含上述3个临床特征和15个影像组学特征的多模态联合模型. 该联合模型在训练集和验证集中的AUC均为0.9458, 联合模型表现优于影像组学模型和临床模型的性能(详见表5, 图4A和B).
| 模型 | 训练集 | 验证集 | ||||
| AUC | 敏感性 | 特异性 | AUC | 敏感性 | 特异性 | |
| 影像组学模型 | 0.8521 | 0.7333 | 0.8750 | 0.8761 | 0.7206 | 0.8750 |
| 临床模型 | 0.9286 | 0.6667 | 0.9167 | 0.9200 | 0.7353 | 0.9062 |
| 多模态联合模型 | 0.9458 | 0.8667 | 1.000 | 0.9357 | 0.7941 | 0.9357 |
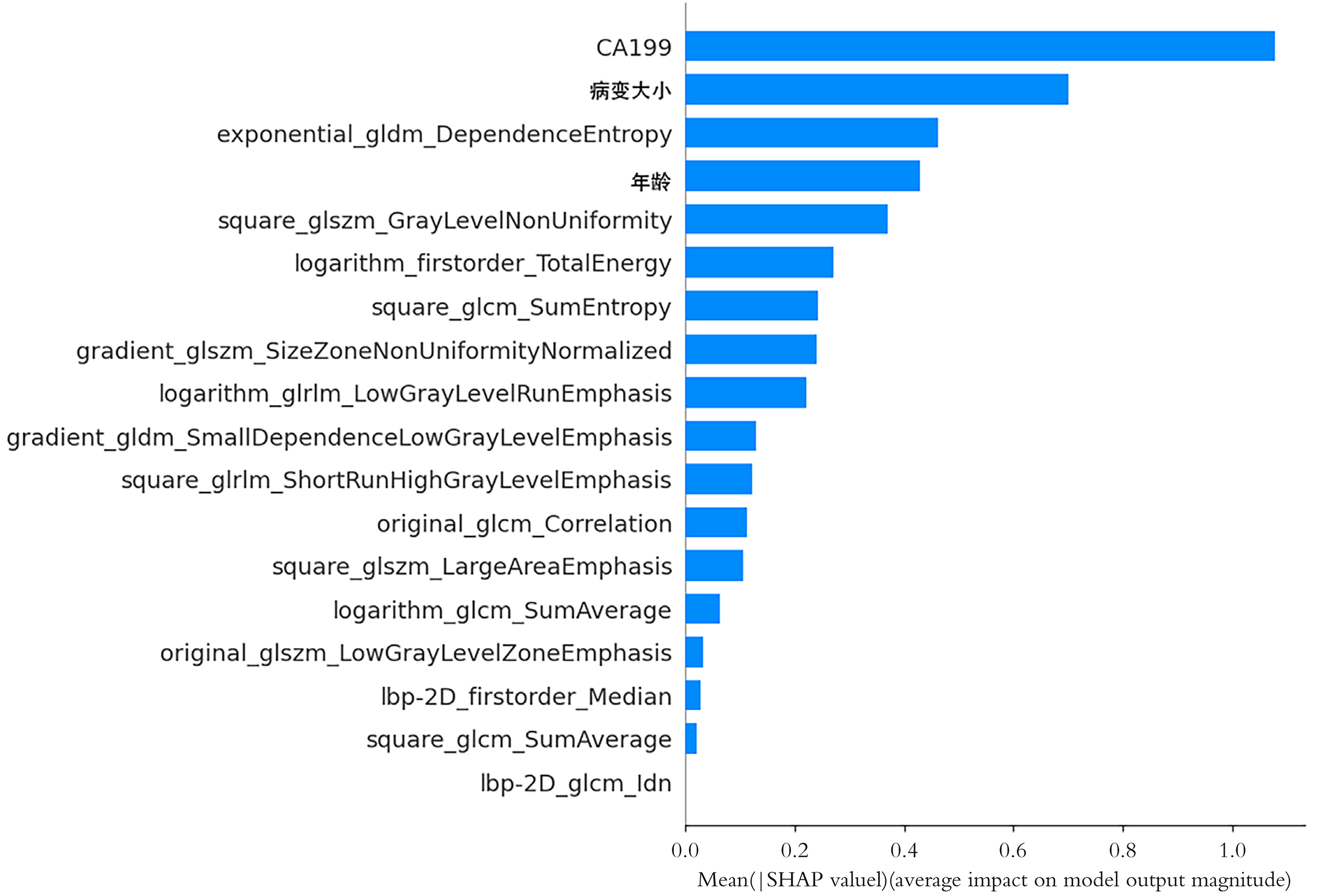
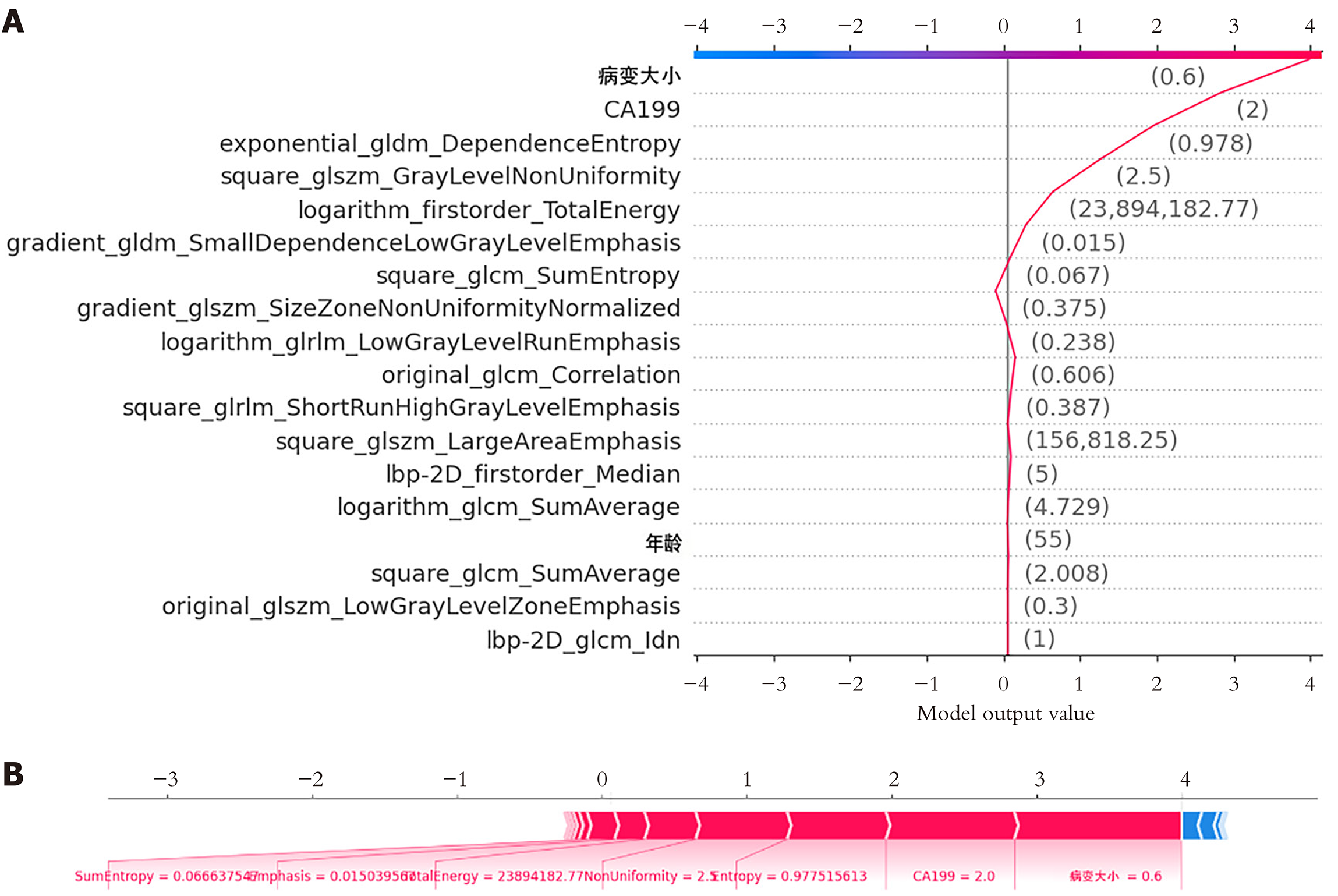
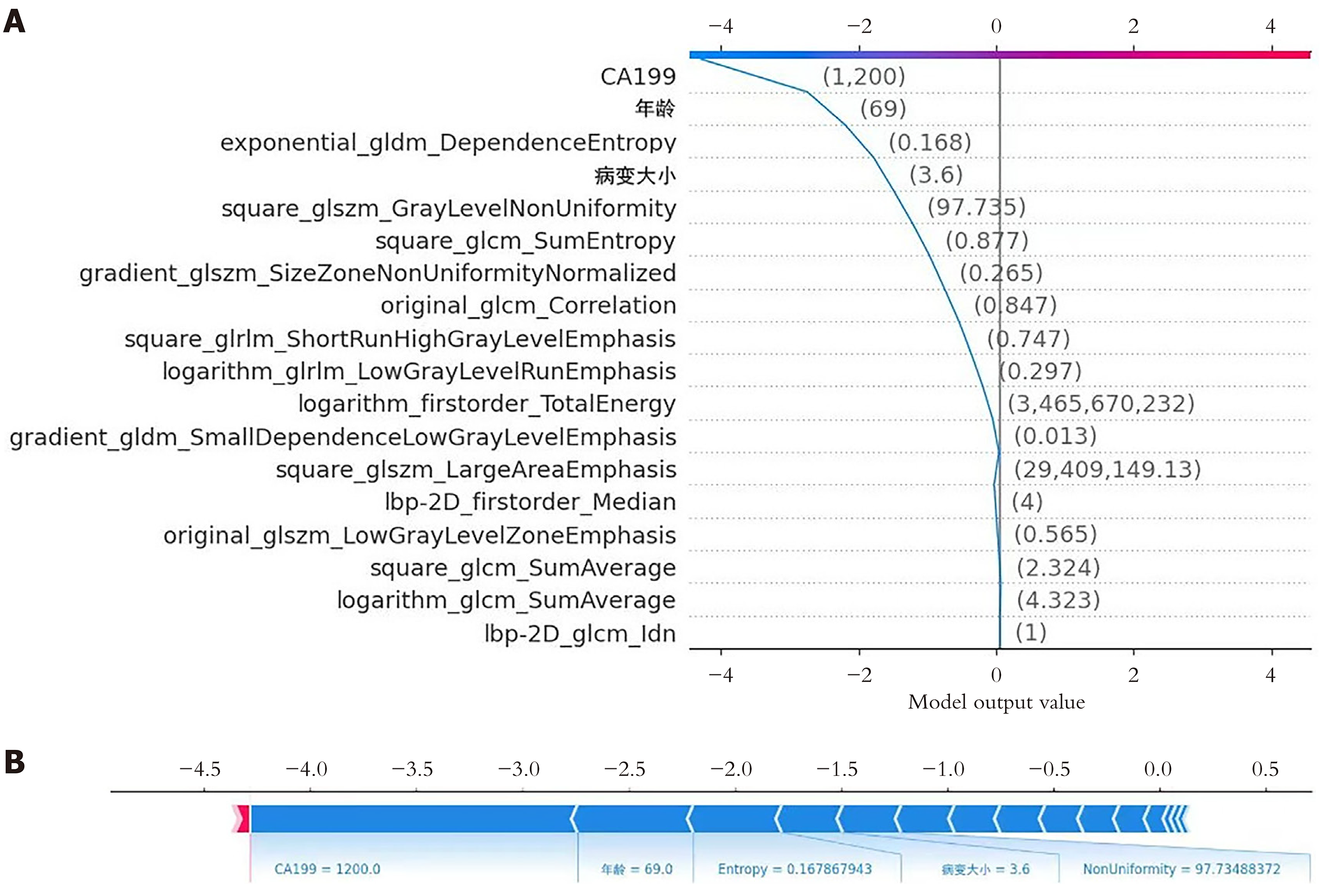
我们采用SHAP方法实现对多模态XGBoost模型特征重要性的概述. SHAP为模型每个特征分配重要性值, 从而帮助识别对给定预测产生最重大影响的关键因素[17]. SHAP条形图(图5)显示了在整体水平上不同特征对联合AI模型预测影响的重要性排序, 其根据平均绝对 SHAP 值从高到低进行排序. 平均绝对SHAP值越高, 对模型预测结果的影响越大. 本研究中对联合XGBoost模型贡献度前5的特征包括3项临床指标及2项影像组学特征. 其中临床指标包括CA199、病变大小、患者年龄, 影像组学特征包括exponential_gldm_DependenceEntropy和square_glszm_GrayLevelNonUniformity, 表明它们与胰腺良恶性诊断密切相关. 同时, SHAP方法除了可在整体队列层面实现对机器学习模型的解释外, 还具有在单个受试者的水平上解释机器学习算法的能力[18]. 因此在本研究中, 我们选取2名代表性样本进行详细的可解释性分析以加强对模型在个人层面决策过程的理解, 如图6和图7. 通过可视化样本的SHAP值, 我们可以判断特征对这些特定样本的预测影响.
本研究通过逐步的特征选择和多模型的比较验证, 确定了在胰腺良恶性病变诊断中有意义的临床实验室指标及影像组学特征, 开发了一种基于XGBoost的多模态机器学习模型来有效地区分胰腺病变的良恶性. 同时使用SHAP方法提供了模型的可解释性. 据我们所知, 这是第一项通过构建多模态ML模型来预测胰腺病变的良恶性的研究.
本研究对训练集的临床指标进行单因素、多因素Logistic回归分析, 结果显示年龄、病变大小及CA199是区分胰腺病变良恶性的独立影响因素(P<0.05). 年龄越大、病变大小越大、CA199水平越高, 胰腺病变恶性概率越大. 既往有研究表明, 胰腺癌的发病率随年龄增长而升高, 90%新诊断的胰腺癌患者年龄在55岁以上, 大多数患者诊断为胰腺癌时的中位年龄为70岁[17,18]. 本研究训练集中胰腺恶性病变患者的中位年龄为63.5岁, 高于胰腺良性病变患者中位年龄(60岁), 表明年龄是影响胰腺癌发病的因素之一. CA199是美国食品药品监督管理局批准用于胰腺癌常规治疗的唯一标志物[19]. 在胰腺肿块患者中, 升高的CA199对胰腺癌有较高的预测价值[20]. 本研究中, 胰腺恶性病变的CA199明显高于胰腺良性病变(467.67 vs 21.15, P<0.001), 表明CA199是影响胰腺癌诊断的因素之一. 先前有研究表明, 胰腺癌的中位大小显著大于胰腺神经内分泌肿瘤[21,22], 本研究与之类似, 胰腺恶性病变的大小大于胰腺良性病变, 差距具有统计学意义(3.85 vs 2.40, P<0.001), 表明病变大小也是胰腺恶性病变的预测因素之一. 综上所述, 本研究中观察到的有意义的临床特征与既往所报道的特征存在一致性, 验证了结果的可靠性.
影像组学通过将传统医学图像转化为可挖掘的高通量特征以分析人类肉眼无法观察到的纹理特征[4], 是一种无创操作. 其对图像的辨别基于算法的客观计算, 非医生的主观判断[23]. 目前, 影像组学在肿瘤学中的应用最成熟, 可提供基于形状、大小或纹理的图像特征以反映肿瘤表型和(或)微环境的信息[24]. Ren等[25]从未增强CT图像中筛选并提取特征, 利用4个最具有预测性的参数生成RF模型, 并进行十次交叉验证, 该模型的平均AUC达0.84; Mukherjee等[26]选择了34个CT影像组学特征构建识别胰腺癌的机器学习模型, 其中SVM的AUC达0.9220; Mo等[22]利用6个非零系数的EUS影像组学特征构ML模型以准确识别胰腺癌和胰腺神经内分泌肿瘤, 其中RF模型在训练集和测试集中AUC分别为0.999和0.649. 本研究通过差异性分析和LASSO回归最终确定了15个影像组学特征, 包括2个一阶特征和13个二阶特征. 其中一阶特征描述图像强度值的分布, 如平均值、最大值、最小值等; 二阶特征描述体素之间的统计关系, 与肿瘤异质性相关[27,28]. 基于上述影像组学特征使用多种主流机器学习算法(GNB、LR、RF、XGBoost和SVC)构建预测模型以区分胰腺病变的良恶性, 以克服单一算法的局限性. 其中XGBoost模型在训练集中AUC为0.8521、准确性为0.8333、敏感性为0.7333、特异性为0.8750、F1分数为0.8000, 在验证集中AUC为0.8761、准确性为0.7955、敏感性为0.7206、特异性为0.8750、F1分数为0.7840, 表现出卓越稳定的性能. XGBoost算法是梯度提升的优化实现, 具有高偏差和低方差的特点. 近年来因其计算速度快、泛化能力强、预测性能高而引起广泛关注[29], 既往有研究也证明了XGBoost模型的高预测性能[30,31], 因此本研究选取XGBoost模型作为我们分析的基础模型. 然而, 仅仅利用上述影像组学特征构建的预测模型在训练集和验证集中的AUC分别为0.8521和0.8761, 预测能力有限. 因此, 我们考虑将有意义的临床特征加入模型以进一步提高预测模型的性能.
Liu等[32]利用243名肝内胆管癌患者的CT影像组学特征和临床放射学特征构建基于机器学习的预测模型, 其中组合XGBoost模型在外部验证和前瞻性队列中的AUC均为0.831, 优于影像组学和临床放射学模型. Guo等[33]利用B超影像组学特征和临床实验室指标构建血吸虫病肝纤维化分级诊断模型, 其中多模态联合SVM模型的AUC在验证集中达0.913, 性能最优. Zhang等[34]根据影像组学特征和临床指标构建ML模型以区分肺纯浸润性粘液腺癌和混合性粘液腺癌, 同样地, 该研究中联合ML模型表现出最佳性能. 但目前尚未发现联合多模态ML模型在预测胰腺良恶性诊断中的应用. 因此, 本研究使用上述15个影像组学参数特征及3个临床特征构建基于XGBoost算法的多模态联合模型, 结果表明多模态联合模型在验证集中AUC为0.9357, 优于临床模型和影像组学模型, 与之前研究结果相似, 表明多模态模型对单影像组学模型具有补充优化能力.
尽管机器学习模型在临床应用中展现出卓越性能, 但其存在"黑匣子"问题, 人类无法理解模型如何做出决策. 因此, 现已出现了多种可解释性人工智能算法来解决该问题. SHAP算法是一种可应用于不同机器学习模型的临床实用的可解释性工具, 其可可视化每个特征的整体或单独贡献, 促进模型的临床应用, 增强临床医生使用预测模型的信心[35]. Gong等研究发现[36], SHAP法与传统的基于权重的解释方法相比, 表现出卓越的一致性和性能. 因此在这项研究中, 我们使用SHAP方法提供ML模型的全局和局部解释, 确定某些特征对模型预测结果的具体贡献, 增加了模型的透明度和可解释性.
本研究构建了多模态模型以预测胰腺病变的良恶性, 有以下三方面优点: 首先与之前研究相比, 该模型结合了EUS影像组学与临床实验室指标多方面信息, 可进一步提高诊断性能; 其次该联合模型与临床工作流程具有一致性. 在临床工作中, 对胰腺病变的诊断不仅仅基于EUS图像, 还要综合临床信息进行综合判断. 因此该模型可通过联合临床实验室指标和EUS图像信息模拟临床决策过程, 更有可能集成到临床工作流程中; 最后我们通过SHAP方法提供了模型的可解释性, 增加医师对模型结果的信任性.
尽管本研究取得上述成果, 但仍存在几个局限性. 首先, 这是一项回顾性研究, 可能存在选择偏倚. 其次, 本研究是单中心研究, 缺乏外部数据验证, 可能会限制研究结果的普遍性. 未来可能需要纳入多中心大样本数据进一步验证模型的稳健性和有效性. 第三, 本研究仅收集临床实验室指标和EUS影像组学特征来构建多模态模型, 未来可进一步纳入病史、CT报告等数据进一步提高诊断性能.
总之, 我们构建了一种基于机器学习的组合模型, 该模型结合了超声内镜影像组学特征和临床特征以有效识别胰腺病变的良恶性. SHAP分析提供了模型的可解释性, 有助于提高临床医生对模型预测结果的接受度和信任度.
超声内镜(endoscopic ultrasonography, EUS)是诊断胰腺病变的一种有价值工具, 但不同医师间的诊断能力差异很大, 限制了EUS诊断的特异性和敏感性. 目前机器学习(machine learning, ML)可通过处理非线性关系、应对复杂的交互以及挖掘大数据中的细微相关性, 从而在预测各种临床结局方面取得了良好的表现. 因此, 本文构建ML模型以提高胰腺良恶性病变的诊断准确性.
本文将利用胰腺病变患者的EUS影像组学特征和临床特征构建首例多模态ML模型以提高临床上胰腺良恶性病变的诊断.
本文旨在构建多模态ML模型以实现胰腺病变的良恶性诊断, 并通过可解释方法克服模型"黑匣子"问题, 帮助临床医师理解接受模型结果.
本文回顾性收集利用了天津医科大学总医院2014-2024年216例胰腺病变患者的临床和超声内镜图像信息. 使用Python 3.8.0和Pyradiomic自动提取经灰度化处理的感兴趣区域中的影像组学特征, 通过t检验和最小绝对收缩和选择运算符逻辑回归算法进行影像组学特征筛选; 通过单因素和多因素Logistic回归进行临床特征筛选. 利用上述影像组学特征开发了5种影像组学机器学习模型: 高斯朴素贝叶斯、逻辑回归、随机森林、支持向量机和极端梯度提升(extreme gradient boosting, XGBoost), 并选取最优XGBoost模型构建多模态联合模型. 沙普利加和解释法(SHapley Additive exPlanations, SHAP)实现模型的可解释性. 据我们所知, 这是首例将临床特征和EUS影像组学特征联合构建可解释机器学习模型以实现胰腺良恶性病变诊断的研究.
本研究构建的多模态联合ML模型在胰腺良恶性病变的诊断中取得优异结果, 其在验证集中曲线下面积达0.9357, 优于临床ML模型和影像组学ML模型. SHAP分析表明对联合XGBoost模型贡献度前5的特征包括3项临床特征及2项影像组学特征.
本研究首次构建联合临床特征和EUS影像组学特征的多模态ML模型以有效识别胰腺病变的良恶性, SHAP分析提供了模型的可解释性, 增加了医师对模型结果的信任.
未来可将病史、CT报告等其他临床信息进一步纳入多模态模型并在多中心进行模型验证以提高模型诊断的准确性及稳健性.
学科分类: 胃肠病学和肝病学
手稿来源地: 天津市
同行评议报告学术质量分类
A级(优秀): A
B级(非常好): B, B
C级(良好): 0
D级(一般): D
E级(差): 0
科学编辑: 刘继红 制作编辑:郑晓梅
| 1. | Nagtegaal ID, Odze RD, Klimstra D, Paradis V, Rugge M, Schirmacher P, Washington KM, Carneiro F, Cree IA; WHO Classification of Tumours Editorial Board. The 2019 WHO classification of tumours of the digestive system. Histopathology. 2020;76:182-188. [PubMed] [DOI] |
| 2. | Bray F, Laversanne M, Sung H, Ferlay J, Siegel RL, Soerjomataram I, Jemal A. Global cancer statistics 2022: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA Cancer J Clin. 2024;74:229-263. [PubMed] [DOI] |
| 3. | Lyu J, Jiang M, Zhu Z, Wu H, Kang H, Hao X, Cheng S, Guo H, Shen X, Wu T, Chang J, Wang C. Identification of biomarkers and potential therapeutic targets for pancreatic cancer by proteomic analysis in two prospective cohorts. Cell Genom. 2024;4:100561. [PubMed] [DOI] |
| 4. | Liu J, Hu L, Zhou B, Wu C, Cheng Y. Development and validation of a novel model incorporating MRI-based radiomics signature with clinical biomarkers for distinguishing pancreatic carcinoma from mass-forming chronic pancreatitis. Transl Oncol. 2022;18:101357. [PubMed] [DOI] |
| 5. | Oettle H, Neuhaus P, Hochhaus A, Hartmann JT, Gellert K, Ridwelski K, Niedergethmann M, Zülke C, Fahlke J, Arning MB, Sinn M, Hinke A, Riess H. Adjuvant chemotherapy with gemcitabine and long-term outcomes among patients with resected pancreatic cancer: the CONKO-001 randomized trial. JAMA. 2013;310:1473-1481. [PubMed] [DOI] |
| 6. | Kennedy T, Preczewski L, Stocker SJ, Rao SM, Parsons WG, Wayne JD, Bell RH, Talamonti MS. Incidence of benign inflammatory disease in patients undergoing Whipple procedure for clinically suspected carcinoma: a single-institution experience. Am J Surg. 2006;191:437-441. [PubMed] [DOI] |
| 7. | Yousaf MN, Chaudhary FS, Ehsan A, Suarez AL, Muniraj T, Jamidar P, Aslanian HR, Farrell JJ. Endoscopic ultrasound (EUS) and the management of pancreatic cancer. BMJ Open Gastroenterol. 2020;7. [PubMed] [DOI] |
| 8. | Yin H, Yang X, Sun L, Pan P, Peng L, Li K, Zhang D, Cui F, Xia C, Huang H, Li Z. The value of artificial intelligence techniques in predicting pancreatic ductal adenocarcinoma with EUS images: A meta-analysis and systematic review. Endosc Ultrasound. 2023;12:50-58. [PubMed] [DOI] |
| 9. | Yang Y, Li L, Qu C, Liang S, Zeng B, Luo Z. Endoscopic ultrasound-guided fine needle core biopsy for the diagnosis of pancreatic malignant lesions: a systematic review and Meta-Analysis. Sci Rep. 2016;6:22978. [PubMed] [DOI] |
| 10. | Huang J, Fan X, Liu W. Applications and Prospects of Artificial Intelligence-Assisted Endoscopic Ultrasound in Digestive System Diseases. Diagnostics (Basel). 2023;13. [PubMed] [DOI] |
| 11. | Hsu WH, Ko AT, Weng CS, Chang CL, Jan YT, Lin JB, Chien HJ, Lin WC, Sun FJ, Wu KP, Lee J. Explainable machine learning model for predicting skeletal muscle loss during surgery and adjuvant chemotherapy in ovarian cancer. J Cachexia Sarcopenia Muscle. 2023;14:2044-2053. [PubMed] [DOI] |
| 12. | Warkentin MT, Al-Sawaihey H, Lam S, Liu G, Diergaarde B, Yuan JM, Wilson DO, Atkar-Khattra S, Grant B, Brhane Y, Khodayari-Moez E, Murison KR, Tammemagi MC, Campbell KR, Hung RJ. Radiomics analysis to predict pulmonary nodule malignancy using machine learning approaches. Thorax. 2024;79:307-315. [PubMed] [DOI] |
| 13. | Stanzione A, Cuocolo R, Verde F, Galatola R, Romeo V, Mainenti PP, Aprea G, Guadagno E, Del Basso De Caro M, Maurea S. Handcrafted MRI radiomics and machine learning: Classification of indeterminate solid adrenal lesions. Magn Reson Imaging. 2021;79:52-58. [PubMed] [DOI] |
| 14. | Yildirim M, Yildirim H. CT radiomics-based machine learning model for differentiating between enchondroma and low-grade chondrosarcoma. Medicine (Baltimore). 2024;103:e39311. [PubMed] [DOI] |
| 15. | Ali S, Akhlaq F, Imran AS, Kastrati Z, Daudpota SM, Moosa M. The enlightening role of explainable artificial intelligence in medical & healthcare domains: A systematic literature review. Comput Biol Med. 2023;166:107555. [PubMed] [DOI] |
| 16. | Yang F, Li C, Yang W, He Y, Wu L, Jiang K, Sun C. Development and validation of an explainable machine learning model for predicting multidimensional frailty in hospitalized patients with cirrhosis. Brief Bioinform. 2024;25. [PubMed] [DOI] |
| 17. | McGuigan A, Kelly P, Turkington RC, Jones C, Coleman HG, McCain RS. Pancreatic cancer: A review of clinical diagnosis, epidemiology, treatment and outcomes. World J Gastroenterol. 2018;24:4846-4861. [PubMed] [DOI] |
| 18. | Cai J, Chen H, Lu M, Zhang Y, Lu B, You L, Zhang T, Dai M, Zhao Y. Advances in the epidemiology of pancreatic cancer: Trends, risk factors, screening, and prognosis. Cancer Lett. 2021;520:1-11. [PubMed] [DOI] |
| 19. | Kim JE, Lee KT, Lee JK, Paik SW, Rhee JC, Choi KW. Clinical usefulness of carbohydrate antigen 19-9 as a screening test for pancreatic cancer in an asymptomatic population. J Gastroenterol Hepatol. 2004;19:182-186. [PubMed] [DOI] |
| 20. | Ballehaninna UK, Chamberlain RS. The clinical utility of serum CA 19-9 in the diagnosis, prognosis and management of pancreatic adenocarcinoma: An evidence based appraisal. J Gastrointest Oncol. 2012;3:105-119. [PubMed] [DOI] |
| 21. | Baxi AC, Jiang Q, Hao J, Yang Z, Woods K, Keilin S, Willingham FF, Cai Q. The effect of solid pancreatic mass lesions on pancreatic duct diameter at endoscopic ultrasound. Endosc Ultrasound. 2017;6:103-108. [PubMed] [DOI] |
| 22. | Mo S, Huang C, Wang Y, Zhao H, Wei H, Qin H, Jiang H, Qin S. Construction and validation of an endoscopic ultrasonography-based ultrasomics nomogram for differentiating pancreatic neuroendocrine tumors from pancreatic cancer. Front Oncol. 2024;14:1359364. [PubMed] [DOI] |
| 23. | Deng Y, Ming B, Zhou T, Wu JL, Chen Y, Liu P, Zhang J, Zhang SY, Chen TW, Zhang XM. Radiomics Model Based on MR Images to Discriminate Pancreatic Ductal Adenocarcinoma and Mass-Forming Chronic Pancreatitis Lesions. Front Oncol. 2021;11:620981. [PubMed] [DOI] |
| 24. | Gillies RJ, Kinahan PE, Hricak H. Radiomics: Images Are More than Pictures, They Are Data. Radiology. 2016;278:563-577. [PubMed] [DOI] |
| 25. | Ren S, Zhao R, Zhang J, Guo K, Gu X, Duan S, Wang Z, Chen R. Diagnostic accuracy of unenhanced CT texture analysis to differentiate mass-forming pancreatitis from pancreatic ductal adenocarcinoma. Abdom Radiol (NY). 2020;45:1524-1533. [PubMed] [DOI] |
| 26. | Mukherjee S, Patra A, Khasawneh H, Korfiatis P, Rajamohan N, Suman G, Majumder S, Panda A, Johnson MP, Larson NB, Wright DE, Kline TL, Fletcher JG, Chari ST, Goenka AH. Radiomics-based Machine-learning Models Can Detect Pancreatic Cancer on Prediagnostic Computed Tomography Scans at a Substantial Lead Time Before Clinical Diagnosis. Gastroenterology. 2022;163:1435-1446.e3. [PubMed] [DOI] |
| 27. | Lohmann P, Bousabarah K, Hoevels M, Treuer H. Radiomics in radiation oncology-basics, methods, and limitations. Strahlenther Onkol. 2020;196:848-855. [PubMed] [DOI] |
| 28. | Liu Z, Wang S, Dong D, Wei J, Fang C, Zhou X, Sun K, Li L, Li B, Wang M, Tian J. The Applications of Radiomics in Precision Diagnosis and Treatment of Oncology: Opportunities and Challenges. Theranostics. 2019;9:1303-1322. [PubMed] [DOI] |
| 29. | Wang K, Tian J, Zheng C, Yang H, Ren J, Liu Y, Han Q, Zhang Y. Interpretable prediction of 3-year all-cause mortality in patients with heart failure caused by coronary heart disease based on machine learning and SHAP. Comput Biol Med. 2021;137:104813. [PubMed] [DOI] |
| 30. | Mao B, Zhang L, Ning P, Ding F, Wu F, Lu G, Geng Y, Ma J. Preoperative prediction for pathological grade of hepatocellular carcinoma via machine learning-based radiomics. Eur Radiol. 2020;30:6924-6932. [PubMed] [DOI] |
| 31. | Xu Q, Lu X. Development and validation of an XGBoost model to predict 5-year survival in elderly patients with intrahepatic cholangiocarcinoma after surgery: a SEER-based study. J Gastrointest Oncol. 2022;13:3290-3299. [PubMed] [DOI] |
| 32. | Liu Z, Luo C, Chen X, Feng Y, Feng J, Zhang R, Ouyang F, Li X, Tan Z, Deng L, Chen Y, Cai Z, Zhang X, Liu J, Liu W, Guo B, Hu Q. Noninvasive prediction of perineural invasion in intrahepatic cholangiocarcinoma by clinicoradiological features and computed tomography radiomics based on interpretable machine learning: a multicenter cohort study. Int J Surg. 2024;110:1039-1051. [PubMed] [DOI] |
| 33. | 郭 照宇, 邵 菊萍, 邹 小青, 赵 琴平, 钱 沛君, 王 文雅, 黄 璐璐, 薛 靖波, 许 静, 杨 坤, 周 晓农, 李 石柱. 基于影像组学与临床实验室指标构建血吸虫病肝纤维化分级诊断模型. 中国血吸虫病防治杂志. 2024;36:251-258. |
| 34. | Zhang J, Hao L, Xu Q, Gao F. Radiomics and Clinical Characters Based Gaussian Naive Bayes (GNB) Model for Preoperative Differentiation of Pulmonary Pure Invasive Mucinous Adenocarcinoma From Mixed Mucinous Adenocarcinoma. Technol Cancer Res Treat. 2024;23:15330338241258415. [PubMed] [DOI] |
| 35. | Ma J, Bo Z, Zhao Z, Yang J, Yang Y, Li H, Yang Y, Wang J, Su Q, Wang J, Chen K, Yu Z, Wang Y, Chen G. Machine Learning to Predict the Response to Lenvatinib Combined with Transarterial Chemoembolization for Unresectable Hepatocellular Carcinoma. Cancers (Basel). 2023;15. [PubMed] [DOI] |
| 36. | Gong K, Lee HK, Yu K, Xie X, Li J. A prediction and interpretation framework of acute kidney injury in critical care. J Biomed Inform. 2021;113:103653. [PubMed] [DOI] |