修回日期: 2015-09-02
接受日期: 2015-09-11
在线出版日期: 2015-10-18
目的: 探索CRISPR干扰(CRISPR interference, CRISPRi)能否实现在体抑制肝脏miR-122表达.
方法: 针对miR-122启动子区设计sgRNA(sgT1和sgT2), 并分别将其与无DNA切割活性仅保留识别活性的dCas9-KRAB载体通过尾静脉流体力学法注射到8-10 wk龄小鼠, 注射1、2、4 wk后通过实时荧光定量PCR(quantitative real-time PCR, qRT-PCR)方法检测肝脏mmu-miR-122的表达; 设计不同的sgRNA浓度梯度, 探索CRISPRi在体抑制肝脏miR-122表达是否存在剂量依赖性; 通过qRT-PCR及Western blot方法检测肝脏miR-122靶分子HOMX1和Cyclin G1的表达变化.
结果: 在注射1 wk和2 wk后, sgT1介导的CRISPRi在体抑制肝脏miR-122的表达水平分别为23%(P<0.05)和16%(P<0.05); 随sgRNA的剂量升高, 肝脏miR-122表达降低, 当lentiGuide-Puro-sgT1质粒为120 μg时, 可将miR-122的表达抑制约30%; CRIPSRi在体抑制肝脏miR-122表达的同时, 上调了miR-122下游靶分子HMOX1和Cyclin G1的表达.
结论: 本研究利用CRISPRi实现了在体抑制肝脏miR-122的表达, 为抗丙型肝炎病毒(hepatitis C virus)的在体治疗提供了新的策略.
核心提示: 本研究利用CRISPRi在体特异性抑制肝脏miR-122的表达, 上调了下游靶分子HMOX1和Cyclin G1的表达, CRISPRi介导的在体基因修饰为抗丙型肝炎病毒(hepatitis C virus)提供了全新的策略.
引文著录: 赵洪礼, 杨景玉, 李桂玲, 毛德华, 李洪运. CRISPRi在体抑制肝脏miR-122的表达. 世界华人消化杂志 2015; 23(29): 4687-4693
Revised: September 2, 2015
Accepted: September 11, 2015
Published online: October 18, 2015
AIM: To investigate whether CRISPRi can repress miR-122 expression in the mouse liver in vivo.
METHODS: sgRNAs (sgT1 and sgT2) were designed, targeting the region of mmu-miR-122 promoter. sgRNAs (sgT1 or sgT2) and catalytically inactive dCas9-KRAB were delivered into 8-10-week-old mice by hydrodynamic tail-vein injection. The expression of mmu-miR-122 was detected at 1, 2 and 4 weeks after injection by quantitative real-time PCR (qRT-PCR) to determine the more effective sgRNA. Different concentrations of sgRNA were tested in order to address whether CRISPRi was concentration dependent in vivo. The expression of miR-122 downstream target genes HOMX1 and CyclinG1 was assessed by qRT-PCR and Western blot.
RESULTS: Compared to the control group, CRISPRi mediated by sgT1 could repress miR-122 expression by 23% (P < 0.05) and 16% (P < 0.05) in the mouse liver at 1 and 2 weeks after injection, respectively. The effect of CRISPRi was enhanced with increased concentrations of sgRNA. After mmu-miR-122 expression in the mouse liver was inhibited by CRISPRi in vivo, the expression of miR-122 downstream target genes HMOX1 and CyclinG1 was upregulated.
CONCLUSION: CRISPRi can specially repress miR-122 expression in the liver in vivo, which provides a novel therapeutic strategy against hepatitis C virus (HCV) infections.
- Citation: Zhao HL, Yang JY, Li GL, Mao DH, Li HY. In vivo inhibition of liver miR-122 expression by CRISPRi. Shijie Huaren Xiaohua Zazhi 2015; 23(29): 4687-4693
- URL: https://www.wjgnet.com/1009-3079/full/v23/i29/4687.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v23.i29.4687
丙型肝炎病毒(hepatitis C virus, HCV)引起的丙型肝炎是一种致命的血液传染病. HCV是RNA病毒, 其高变异性使其得以避过免疫系统, 尽管聚乙二醇化干扰素和利巴韦林这类抗病毒药物具有一定的疗效, 然而HCV的高变异性, 导致耐药性病毒突变体的产生[1-3]. 研究[4,5]发现, 肝脏特异性表达的微小RNA-122(microRNA-122, miR-122)与HCV病毒的生长和复制有关, 当miR-122失去活性时, HCV的RNA复制降低约80%. miR-122属于小片段非编码RNA, miR-122与HCV RNA的5'端非翻译区(5' untranslated region, 5'UTR)的两个靶位点结合, 形成miR-122-HCV复合体, 保护HCV RNA不被核酸内切酶水解或者不被宿主免疫反应清除[5,6]. miR-122与HCV RNA的结合位点高度保守, 这使得miR-122成为抗HCV病毒治疗的宿主靶点, 如果有效抑制肝脏内miR-122的表达, 将有助于降低HCV数量, 为丙型肝炎的治疗提供全新的策略.
CRISPR(clustered regularly interspaced short palindromic repeat sequences)/CRISPR相关基因(CRISPR-associated genes, Cas)系统是一种防御系统, 用以保护细菌和古细菌细胞不受病毒的侵害. 2012年, Jinek等[7]首先利用CRISPR/Cas9系统对外源DNA靶序列进行了精确切割, 从此CRISPR/Cas9系统成为最高效的基因编辑技术. RuvC1结构域及HNH结构域是Cas9的2种核酸酶结构域, HNH结构域切割与sgRNA互补的模板链, RuvC1结构域对另一条链进行切割[8]. Qi等[9]将CRISPR系统改造为特异性抑制靶分子表达的CRISPR干扰(CRISPR interference, CRISPRi)体系: 即将Cas9的核酸内切酶结构域突变(RuvC1, D10A; HNH, H841A), 产生dCas9, 只有DNA识别能力, 没有内切酶活性, 当dCas9与sgRNA共表达时, 形成识别特定位点的DNA复合体, 该复合体可以特异性的干扰RNA聚合酶结合或者转录因子结合, 有效抑制靶分子的表达. 2014年, Yin等[10]首次利用CRISPR/Cas9系统实现了在体基因编辑, 该研究将CRISPR/Cas9系统各个组分经尾静脉高压注射到小鼠体内, 最终约1/250的肝细胞表达了野生型Fah蛋白, Fah阳性肝细胞恢复或者延缓了小鼠体质量的下降. 同年, Xue等[11]利用CRISPR/Cas9系统对野生型小鼠进行在体基因编辑产生癌症模型, 即将表达Cas9的质粒以及靶向肿瘤抑制因子Pten和/或P53的sgRNA注射到小鼠肝脏, 最终产生了与Cre-Loxp系统产生的基因敲除小鼠表型相似的基因突变小鼠. 上述两项研究为CRISPR/Cas9系统的在体基因治疗提供了全新的理念. 本研究探索了CRISPRi在体特异性抑制肝脏miR-122的表达, CRISPRi介导的在体基因修饰为抗HCV病毒提供了全新的策略.
lentiGuide-Puro质粒(#52963)、dCas9-KRAB质粒(#46911)购自Addgene; 载体构建用的BsmBⅠ酶(NEB, R0580S)、碱性磷酸酶FastAP(#EF0651)、T4连接酶(NEB, M0202)、核酸纯化试剂盒(EasyPure Quick Gel Extraction Kit, EG101-01)、反转录试剂盒(EasyScript First-Strand cDNA Synthesis SuperMix, AE301-02)购自北京全式金生物公司; 引物由上海生工合成; polyA聚合酶(NEB, M0276S), TRIzol(Invitrogen, 15596-026), SYBR Premix Ex Tag试剂盒(TaKaRa, DRR041A), anti-Cyclin G1一抗(sc-7865, Santa Cruz), anti-HMOX1一抗(Abcam cat. ab137749); anti-β-actin一抗(Abcam cat. 6276), RIPA Buffer裂解液、考马斯亮蓝(R-250)购自碧云天生物; Western blot显色液(SuperSignal™ West Pico Chemiluminescent Substrate)购自Thermo公司.
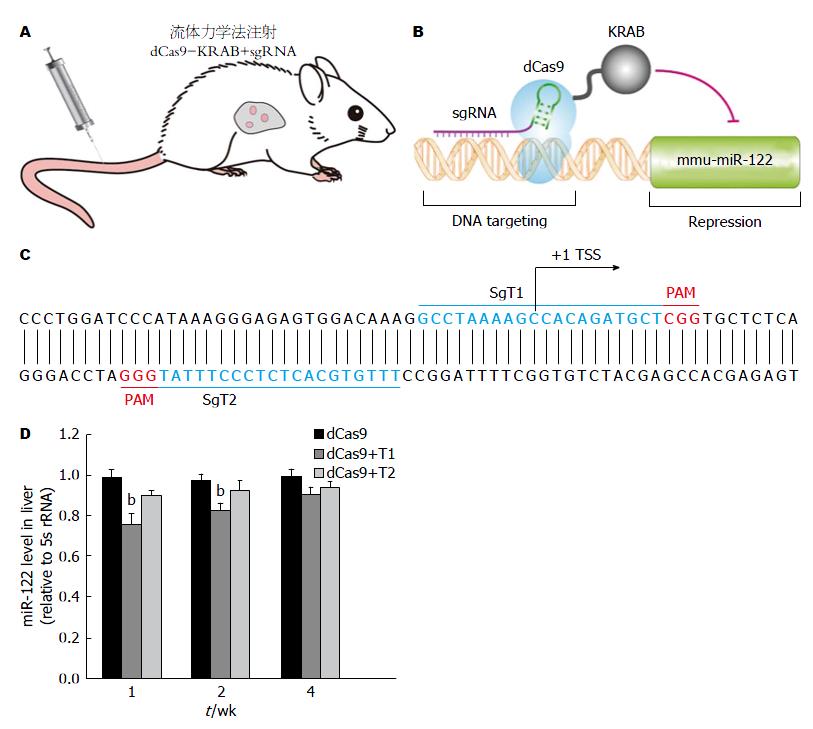
1.2.1 载体构建: 靶向mmu-miR-122启动子区sgRNA载体的构建: 采用BsmBⅠ酶将lentiGuide-Puro载体线性化, 并通过碱性磷酸酶FastAP去磷酸化, 利用EasyPure Quick Gel Extraction Kit进行核酸纯化. 将合成的sgT1及sgT2(sgT1-F和sgT1-R, sgT2-F和sgT2-R, 序列如)分别进行退火与磷酸化, 并与线性化的lentiGuide-Puro载体进行连接, 分别构建分别靶向mmu-miR-122启动子区的sgRNA载体lentiGuide-Puro-sgT1和lentiGuide-Puro-sgT2.
1.2.2 小鼠尾静脉流体力学法注射: 将混合质粒流体力学法注射入小鼠尾静脉(图1A), 即dCas9-KRAB(60 μg)与lentiGuide-Puro(空载体, 60 μg)或lentiGuide-Puro-sgT1(60 μg)或lentiGuide-Puro-sgT2(60 μg), 溶解于2 mL生理盐水, 4-7 s内注射入8-10 wk龄小鼠尾静脉, 1、2、4 wk后分别取小鼠肝脏, 用于检测肝脏miR-122的表达; 流体力学法注射混合质粒, 即dCas9-KRAB(60 μg)与lentiGuide-Puro(空载体)或不同剂量的lentiGuide-Puro-sgT1(30、60或120 μg), 1 wk后分别取小鼠肝脏, 用于检测肝脏miR-122的表达, 并检测高剂量(120 μg)组miR-122靶分子HMOX1、Cyclin G1 mRNA及蛋白表达.
1.2.3 总RNA提取及反转录: (1)总RNA提取: 用1 mL TRIzol裂解肝脏组织(约10 mg), 室温放置5 min, 加入0.2 mL氯仿, 盖紧管盖, 手动剧烈震摇15 s, 然后室温静置2-3 min, 4 ℃ 12000 g离心15 min, 小心吸取上层水相加入到新EP管中, 加入0.5 mL异丙醇颠倒混匀, 室温静置10 min, 4 ℃, 12000 g离心10 min, 后弃上清, 加入1 mL 75%乙醇(DEPC H2O配制)润洗, 4 ℃, 7000 g离心5 min, 弃上清, 将RNA沉淀溶解于适量的无RNase水中, 吸出少量测定浓度, RNA溶液可用于进一步实验, 或者-80 ℃保存; (2)反转录: 取1 μg RNA, 加入1 μL Oligo(dT)20, 无RNase水配成10 μL体系, 65 ℃ 5 min进行变性, 后立即置于冰上, 随后加入2 μL dNTP Mixture、1 μL RNase Inhibitor、1 μL反转录酶以及Buffer和水, 配成20 μL体系, 42 ℃ 1 h, 99 ℃ 5 min, 4 ℃ 5 min, 瞬时离心, -20 ℃保存, 以备实时荧光定量PCR(quantitative real-time PCR, qRT-PCR)使用.
1.2.4 miRNA定量检测: 采用polyA聚合酶加A法[12,13]检测miR-122的表达. (1)加A反应: 取1 μg RNA, 加入1 μL ATP、1 μL PolyA polymerase以及Buffer和无RNase水, 配成10 μL体系, 37 ℃ 1 h, 65 ℃ 20 min, 4 ℃ 5 min; (2)反转录: 加A后, 加入1 μL RNase Inhibitor、1 μL反转录酶、0.5 μL Poly(T) adapter (10 μM)[序列为GCGAGCACAGAATTAATACGACTCACTATAGG(T)12VN]、2 μL dNTP Mixture以及Buffer和水, 配成20 μL体系, 42 ℃1 h, 99 ℃ 5 min, 4 ℃ 5 min; (3)qRT-PCR检测miR-122.
1.2.5 qRT-PCR: 使用SYBR Premix Ex Tag试剂盒, 引物序列如, 使用ABI 7500仪器, 采用两步法PCR反应程序, Stage1: 预变性, 95 ℃ 30 s; Stage2: PCR反应(40 cycles): 95 ℃ 5 s, 60 ℃ 34 s; Stage3(解离stage): 95 ℃ 15 s, 60 ℃ 1 min, 95 ℃ 15 s, 后进行数据分析.
1.2.6 Western blot: 利用RIPA Buffer裂解肝脏组织提取总蛋白, 考马斯亮蓝(R-250)测定蛋白浓度, 上样20 μg, 电泳, 转膜, 奶粉封闭, 一抗孵育4 ℃过夜, 洗膜, 二抗孵育(HRP标记, 1:10000), 洗膜, 显色, 采集图像.
统计学处理 采用SPSS12.0统计学处理软件进行t检验分析, 各项检测结果以mean±SD记录. 以P<0.05为差异具有统计学意义.
本研究利用小鼠尾静脉流体力学法将表达dCas9及靶向miR-122启动子区的sgRNA的质粒注射到小鼠体内, dCas9仅保留DNA识别能力而无内切酶活性, dCas9与KRAB(kruppel-associated box)转录抑制结构域融合, 利用CRISPRi原理特异性抑制miR-122的转录(图1A, B). 我们设计了2个靶向miR-122启动子区sgRNA, 命名为T1和T2(图1C). 研究结果显示, 注射1 wk和2 wk后, sgT1介导的CRISPRi显著性降低肝脏miR-122的表达, 但是, 注射4 wk后, 抑制效果下降, 与对照组没有显著性差异(图1D). 研究结果揭示, CRISPRi可以在体抑制肝脏miR-122的表达.
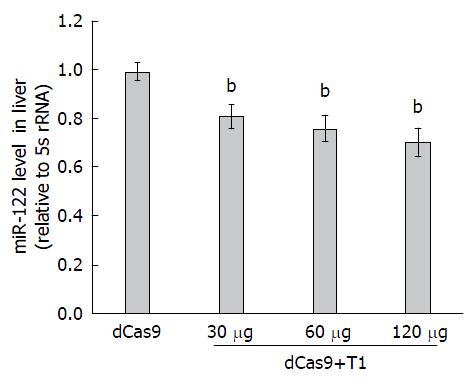
在前期结果的基础上, 进一步探索了sgRNA剂量对miR-122抑制效果的影响. 我们将检测时间定为抑制效果较为明显的1 wk, 分别采用30、60和120 μg的质粒lentiGuide-Puro-sgT1. 研究结果表明, 随着sgRNA浓度的增加, CRISPRi对miR-122的抑制作用提高. 与对照组相比, 当lentiGuide-Puro-sgT1质粒为120 μg时, 可将miR-122的表达抑制约30%(图2). 因此, CRIPSRi在体抑制肝脏miR-122表达依赖于sgRNA的剂量, sgRNA剂量越高, 抑制效果提高.
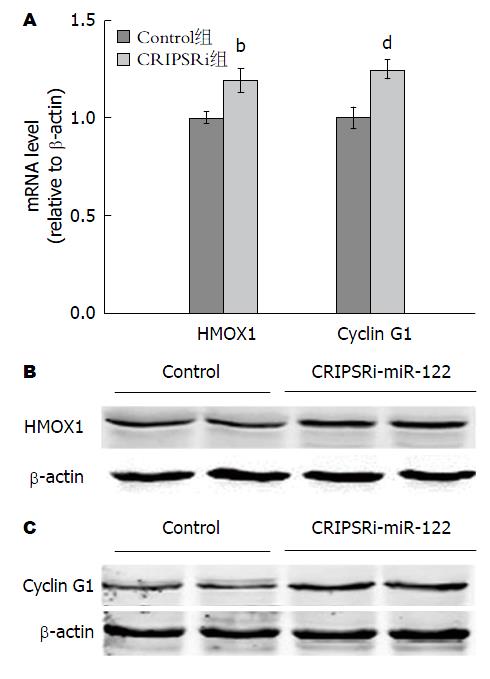
HMOX1和Cyclin G1在HCV复制中发挥抑制作用, 而miR-122通过下调HMOX1和Cyclin G1的表达, 而间接促进HCV复制[14,15]. 为探索CRIPSRi在体抑制肝脏miR-122表达的同时, 能否进一步实现对miR-122下游靶分子的表达调控, 本研究在共同注射dCas9-KRAB和lentiGuide-Puro-sgT1(120 μg)后, 检测了肝脏的HMOX1和Cyclin G1的mRNA及蛋白表达, 结果显示(图3), 注射1 wk后, 肝脏HMOX1 mRNA和Cyclin G1 mRNA水平均显著升高, 同时HMOX1和Cyclin G1蛋白水平也有明显升高. 因此, CRIPSRi在体抑制肝脏miR-122表达的同时, 并上调肝脏miR-122下游靶分子HMOX1和Cyclin G1的表达.
本研究首次利用CRIPSRi实现在体抑制肝脏miR-122表达, 并且上调miR-122下游靶分子HMOX1和Cyclin G1的表达, 为CRISPRi应用于抗HCV病毒的在体治疗提供了理论依据. CRISPR/Cas系统广泛存在于细菌及古生菌中, 是机体长期进化中形成的RNA指导的适应性免疫系统, 用于降解入侵病毒或噬菌体DNA. 2012年, Jinek等[7]首先利用CRISPR/Cas9系统对外源DNA靶序列进行了精确修饰, 开辟了RNA介导的基因编辑时代. 目前, CRISPR/Cas9系统已被广泛应用于细胞及动物的基因编辑, 包括人细胞、小鼠、大鼠、斑马鱼及食蟹猴等. Qi等[9]将CRISPR/Cas9改造为CRISPRi体系, CRISPRi通过干扰DNA的转录实现对靶分子的表达抑制. Gilbert等[16]研究发现, CRISPRi可分别有效抑制人类细胞系及酵母靶分子的表达5-15倍及50倍. 此外, Gilbert等还发现CRISPRi在高效抑制靶分子表达的同时, 脱靶率极低; 由于CRISPRi作用于DNA水平, CRISPRi可应用于非编码RNA、核定位的RNA、转录物的反义序列以及聚合酶Ⅲ转录产物[16]. RNAi是作用于RNA水平的基因转录抑制的方法[17]. RNAi和CRISPRi在许多研究中成为互补的研究方法, 然而CRISPRi更具有潜在的优势. 锁核酸标记的miravirsen(SPC3469)作为新的抗HCV宿主靶向药物, 他具有较高的基因屏障、泛基因型的抗病毒活性, 将应用于丙型肝炎临床治疗. 但是, 相对于锁核酸标记的寡核苷酸成本高、易降解的特点, 利用CRISPRi具有其自身的优势: 成本低、易操作、高效. 本研究组前期利用CRISPRi实现了HepG2细胞内miR-122的特异性表达抑制. Yin等[10]利用CRISPR/Cas9系统实现了在体基因编辑, 该研究将CRISPR/Cas9系统各个组分经尾静脉流体力学注射到小鼠体内, 最终约1/250的肝细胞表达了野生型Fah蛋白, Fah阳性肝细胞恢复或者延缓了小鼠体质量的下降. Xue等[11]利用CRISPR/Cas9系统对野生型小鼠进行在体基因编辑进而产生癌症模型, 将表达Cas9的质粒以及靶向肿瘤抑制因子Pten的sgRNA(sgPten)注射到小鼠, 注射2 wk后, 检测到肝脏Pten阴性细胞; 注射4 mo后, 肝细胞内出现明显的脂质积累, 以及AKT磷酸化水平提高, 这与Cre-Loxp系统产生的Pten基因敲除小鼠模型类似; 而同时靶向Pten和P53的sgRNA(sgPten和sgP53)诱导的肝脏肿瘤与Cre-Loxp系统介导的Pten和P53基因敲除表型类似; 肝脏和肿瘤组织的DNA测序结果显示, 肿瘤抑制基因Pten和P53存在插入和缺失突变, 同时包括肿瘤中Pten和P53的双等位基因突变. Xue等[11]还探索了CRISPR/Cas9系统直接介导在体获得性功能突变, 将Cas9质粒与靶向Ctnnb1(编码β-catenin)的sgRNA以及200bp的单链DNA(含4个点突变, 突变后的β-catenin将更加稳定, 并且定位于细胞核)供体共同注射野生型小鼠, 注射后7 d, 部分肝细胞出现核定位表达的β-catenin[11]. 结合CRISPR/Cas9系统在体基因编辑功能以及前期研究基础, 我们探索发现CRISPRi可在体抑制肝脏miR-122表达, 同时, miR-122下游靶分子HMOX1和Cyclin G1均表达升高, 这为CRISPRi应用于抗HCV病毒的在体治疗提供了全新的策略.
丙型肝炎病毒(hepatitis C virus, HCV)可诱发丙型肝炎, 导致肝硬化和肝癌等疾病. 然而到目前为止仍没有一种有效的治疗方式. 微小RNA-122(microRNA-122, miR-122)作为HCV复制的必需元件, 有可能成为治疗丙型肝炎的新的抗病毒靶点.
毛高平, 教授, 中国人民解放军解放军空军总医院
miR-122是宿主细胞的内源性分子, 为HCV病毒复制的必需元件. 高效且特异抑制miR-122成为丙型肝炎治疗的关键.
Lanford等利用miR-122的反义锁核酸miravirsen(SPC3469)抑制HCV病毒的复制, 开启了靶向miR-122治疗丙型肝炎的先河. 本课题组首次利用CRISPR干扰(CRISPR interference, CRISPRi)技术实现了对肝癌细胞HepG2内源性miR-122表达的抑制.
CRISPRi技术因特异性强, 成本低, 易操作, 高效等特点, 成为极具前景的基因治疗方式. 本研究首次利用CRISPRi技术在体抑制了miR-122的表达, 为抗HCV病毒提供了新的思路, 因而具有广阔的应用前景.
CRISPRi可在体抑制miR-122表达从而达到抗HCV病毒的目的, 为分子靶向药物治疗丙型肝炎提供了理论基础.
CRISPRi: 天然CRISPR相关基因9(CRISPR-associated gene 9, Cas9)含有2种核酸酶结构域, RuvC1结构域及HNH结构域, HNH结构域切割与crRNA互补的模板链, RuvC1结构域切割非互补链. 将Cas9的核酸内切酶结构域突变(RuvC1, D10A; HNH, H841A)产生dCas9, 仅保留DNA识别能力而无内切酶活性, 当dCas9与sgRNA共表达时, 形成识别特定位点的复合体, 该复合体可以特异性的干扰RNA聚合酶或者转录因子与DNA序列的结合, 有效抑制靶分子的表达, 将此过程称为CRISPRi.
本文选题较新, 设计合理, 实验方法先进, 结果有新意, 对于丙型肝炎的抗病毒治疗在理论和实践上均有参考价值. 论文内容虽是临床医生较为生疏的基础实验室工作, 但结合临床, 文字表述较为清晰.
| 1. | Poordad F, McCone J, Bacon BR, Bruno S, Manns MP, Sulkowski MS, Jacobson IM, Reddy KR, Goodman ZD, Boparai N. Boceprevir for untreated chronic HCV genotype 1 infection. N Engl J Med. 2011;364:1195-1206. [PubMed] [DOI] |
| 2. | Sherman KE, Flamm SL, Afdhal NH, Nelson DR, Sulkowski MS, Everson GT, Fried MW, Adler M, Reesink HW, Martin M. Response-guided telaprevir combination treatment for hepatitis C virus infection. N Engl J Med. 2011;365:1014-1024. [PubMed] [DOI] |
| 3. | Bandiera S, Pfeffer S, Baumert TF, Zeisel MB. miR-122--a key factor and therapeutic target in liver disease. J Hepatol. 2015;62:448-457. [PubMed] [DOI] |
| 4. | Henke JI, Goergen D, Zheng J, Song Y, Schüttler CG, Fehr C, Jünemann C, Niepmann M. microRNA-122 stimulates translation of hepatitis C virus RNA. EMBO J. 2008;27:3300-3310. [PubMed] [DOI] |
| 5. | Jopling CL, Yi M, Lancaster AM, Lemon SM, Sarnow P. Modulation of hepatitis C virus RNA abundance by a liver-specific MicroRNA. Science. 2005;309:1577-1581. [PubMed] |
| 6. | Machlin ES, Sarnow P, Sagan SM. Masking the 5' terminal nucleotides of the hepatitis C virus genome by an unconventional microRNA-target RNA complex. Proc Natl Acad Sci U S A. 2011;108:3193-3198. [PubMed] [DOI] |
| 7. | Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 2012;337:816-821. [PubMed] [DOI] |
| 8. | Doudna JA, Charpentier E. Genome editing. The new frontier of genome engineering with CRISPR-Cas9. Science. 2014;346:1258096. [PubMed] [DOI] |
| 9. | Qi LS, Larson MH, Gilbert LA, Doudna JA, Weissman JS, Arkin AP, Lim WA. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 2013;152:1173-1183. [PubMed] [DOI] |
| 10. | Yin H, Xue W, Chen S, Bogorad RL, Benedetti E, Grompe M, Koteliansky V, Sharp PA, Jacks T, Anderson DG. Genome editing with Cas9 in adult mice corrects a disease mutation and phenotype. Nat Biotechnol. 2014;32:551-553. [PubMed] [DOI] |
| 11. | Xue W, Chen S, Yin H, Tammela T, Papagiannakopoulos T, Joshi NS, Cai W, Yang G, Bronson R, Crowley DG. CRISPR-mediated direct mutation of cancer genes in the mouse liver. Nature. 2014;514:380-384. [PubMed] [DOI] |
| 12. | Shi R, Chiang VL. Facile means for quantifying microRNA expression by real-time PCR. Biotechniques. 2005;39:519-525. [PubMed] |
| 13. | Carlsbecker A, Lee JY, Roberts CJ, Dettmer J, Lehesranta S, Zhou J, Lindgren O, Moreno-Risueno MA, Vatén A, Thitamadee S. Cell signalling by microRNA165/6 directs gene dose-dependent root cell fate. Nature. 2010;465:316-321. [PubMed] [DOI] |
| 14. | Shan Y, Zheng J, Lambrecht RW, Bonkovsky HL. Reciprocal effects of micro-RNA-122 on expression of heme oxygenase-1 and hepatitis C virus genes in human hepatocytes. Gastroenterology. 2007;133:1166-1174. [PubMed] |
| 15. | Hou W, Bukong TN, Kodys K, Szabo G. Alcohol facilitates HCV RNA replication via up-regulation of miR-122 expression and inhibition of cyclin G1 in human hepatoma cells. Alcohol Clin Exp Res. 2013;37:599-608. [PubMed] [DOI] |
编辑: 于明茜 电编:都珍珍