修回日期: 2010-03-01
接受日期: 2010-03-02
在线出版日期: 2010-03-28
目的: 研究抗菌药物使用后, 引起腹泻患者肠道杆菌产超广谱β-内酰胺酶(ESBLs)细菌的耐药基因型并进行序列分析.
方法: 收集临床住院应用抗菌药物后出现腹泻患者的大便或肛拭子进行培养, 分离产ESBLs的肺炎克雷伯菌50株、大肠埃希菌21株和变形杆菌18株, 通过改良Hodge实验、EDTA双纸片增效试验、分别测定KPC、金属酶; 聚合酶链反应(PCR)检测其耐药基因、分析PCR产物序列并在GenBank中比对分析, 总结产ESBLs细菌同源性的和变异情况.
结果: 89株产ESBL的菌株中, 有64株携带CTX型耐药基因, 68株携带TEM型耐药基因, 21株携带SHV型耐药基因. 肺炎克雷伯菌中CTX型占到84%(42/50), TEM型占到76%(38/50), SHV型占到15%(7/50); 变形杆菌中没有SHV型, 而TEM型占到100%(18/18), CTX型占到40%(7/18); 大肠埃希菌中CTX占到100%(21/21), SHV型和TEM型均为83%(17/21); 同时检出3株产KPC和22株产金属酶的细菌.
结论: 本院抗生素腹泻患者肠道杆菌产ESBLs耐药基因主要以CTX和TEM为主, 同时携带两个或两个以上耐药基因的细菌占49%(44/89).
引文著录: 冯羡菊, 张燕, 罗予, 郑鹏远. 抗生素性腹泻肠道杆菌产超广谱β-内酰胺酶基因型的调查. 世界华人消化杂志 2010; 18(9): 883-888
Revised: March 1, 2010
Accepted: March 2, 2010
Published online: March 28, 2010
AIM: To genotype extended-spectrum β-lactamase (ESBL)-producing enterobacteriaceae isolated from patients with antibiotic-associated diarrhea.
METHODS: The feces samples were collected from patients with antibiotic-associated diarrhea and cultured to isolate ESBL-producing enterobacteriaceae. Klebsiella pneumoniae carbapenemase production was evaluated by the modified Hodge test. Metallo-β-lactamase production was determined by the imipenem-EDTA double-disk synergy test. Antibiotic resistance genes were amplified by polymerase chain reaction (PCR). The sequences of PCR products were determined and compared with those deposited in GenBank.
RESULTS: Fifty Klebsiella pneumoniae strains, 21 Escherichia coli strains and 18 Proteus strains were isolated. Of all isolated strains, 64 carry ESBLs of the CTX-type, 68 carry ESBLs of the TEM-type, and 21 carry ESBLs of the SHV-type. CTX-, TEM- and SHV-type ESBLs were present in 84% (42/50), 76% (38/50) and 15% (7/50) of Klebsiella pneumoniae isolates, respectively. TEM- and CTX-type ESBLs were present in 100% (18/18) and 40% (7/18) of Proteus isolates, respectively. No SHV-type ESBLs were detected in Proteus isolates. CTX-, TEM- and SHV-type ESBLs were present in 100% (21/21), 83% (17/21) and 83% (17/21) of Escherichia coli isolates, respectively.
CONCLUSION: The enterobacteriaceae isolated from patients with antibiotic-associated diarrhea in our study mainly carry TEM- and CTX-type ESBLs.
- Citation: Feng XJ, Zhang Y, Luo Y, Zheng PY. Genotyping of extended-spectrum β-lactamase-producing enterobacteriaceae isolated from patients with antibiotic-associated diarrhea. Shijie Huaren Xiaohua Zazhi 2010; 18(9): 883-888
- URL: https://www.wjgnet.com/1009-3079/full/v18/i9/883.htm
- DOI: https://dx.doi.org/10.11569/wcjd.v18.i9.883
由于抗生素大量、甚至盲目使用, 临床在治疗感染性疾病的同时, 使肠道中有益菌被抑制, 肠道的微生态平衡被打破, 导致菌群失调, 此时肠上皮细胞的代谢调节水平和生理功能发生紊乱, 使腹泻进一步加重, 即产生抗生素相关性腹泻[1]. 抗生素性腹泻、肠道菌群失调症、细菌的耐药等问题给患者带来了经济、肉体上的痛苦. 自1983年德国学者首先在肺炎克雷伯菌中发现了超广谱β-内酰胺酶(ESBLs)[2], 由于这种酶能够水解广谱头孢菌素, 近年来人们对于他的基因分型、耐药机制及传播方式的研究越来越多. 一般认为, ESBLs大多源于TEM、SHV位的基因突变. 有关抗生素相关性腹泻患者的肠道杆菌产ESBLs分型情况报道的很少. 本文对住院患者因各种感染使用抗菌药物后导致腹泻的大便进行培养, 分离出产ESBLs的肺炎克雷伯菌、大肠埃希菌和变形杆菌做耐药基因型和流行趋势及序列分析, 结果如下.
收集2009年郑州大学第一附属医院临床科室随机病例, 因非肠道感染使用抗菌药物后出现轻微或严重腹泻, 取其不重复患者大便126份进行培养, 他们均曾使用过头孢菌素类、喹诺酮类、碳青霉烯类等两种或两种以上抗菌药物. 质控菌株为大肠埃希菌ATCC25922(产酶)、ATCC35218(不产酶)、肺炎克雷伯菌ATCC700603; ATB细菌鉴定仪, 法国BioMerieux公司; 药敏纸片、头孢噻肟、头孢噻肟/克拉维酸、头孢他啶、头孢他啶/克拉维酸, 厄他培南、美罗培南, 英国Oxoid公司产品; PCR扩增仪(Biometra TGRADIENT); PCR MIX、标记物, 天根生化科技有限公司; 电泳及凝胶成像系统(BIO-RAD); 凝胶纯化及测序, 大连宝生物有限公司; API 20E板条, 法国BioMerieux公司产品.
1.2.1 耐药表型检测: (1)ESBLs检测: 常规K-B纸片扩散法, 选择头孢噻肟(30 μg)、头孢噻肟/克拉维酸(30 μg/10 μg); 头孢他啶(30 μg)、头孢他啶/克拉维酸(30 μg/10 μg)两组进行ESBL表型确证试验. 以CLSI2009版判定标准[8], 任何一组复合剂纸片抑菌环直径大于或等于单独抗生素纸片抑菌环直径5 mm, 即判定为产ESBL菌株, 并将其进行基因分型; (2)改良Hodge实验: 将0.5个麦氏单位ATCC25922做10倍稀释, 涂于MH平板上. 在平板中心贴厄他培南纸片, 将待检菌和阳性对照菌分别按2009版CLSI方法接种至平板. 35 ℃培养过夜, 取出观察; (3)金属碳青霉烯酶检测0.5麦氏单位的待测菌涂布MH平板, 分别贴美罗培南的纸片和含8 μL 0.5 mol/L EDTA-Na2的美罗培南纸片, 纸片间距离大于15 mm. 35℃过夜培养, 观察美罗培南和含EDTA-Na2美罗培南纸片对细菌的抑制情况, 后者与前者抑菌环直径相差>7 mm的为金属酶阳性.
1.2.2 耐药基因检测: CTX型、SHV型和TEM型β-内酰胺酶基因; KPC-2型碳青霉烯酶基因及金属酶等基因表型及引物见相关文献(表1), 进行PCR扩增, 所用引物均由大连宝生物有限公司合成. PCR反应后经产物纯化、测序, 序列在GenBank上检索比对, 确认表型. 分别与参考序列EU938349.1、GQ343054.1、GU064388.1比对. 反应体系: 缓冲液5 μL, dNTP 1 μL, 引物各0.5 μL, 聚合酶0.25 μL, 模板2 μL. 反应条件: 94 ℃, 5 min, 94 ℃, 1 min, 55 ℃, 1 min, 72 ℃, 1 min, 72 ℃, 7 min, 4 ℃保存. 100 V电泳20 min.
| 目标基因 | 引物序列(5'-3') | 大小(bp) | |
| CTX[4] | F: ATG TGC AGY ACC AGT AAR GT | 593 | |
| R: TGG GTR AAR TAR GTS ACC AGA | |||
| TEM[5] | F: TCA ACA TTT CCG TGT CG | 860 | |
| R: CTG ACA GTT ACC AAT GCT TA | |||
| SHV[5] | F: ATG CGT TAT ATT CGC CTG TG | 780 | |
| R: AGA TAA ATC ACC ACA ATG CGC | |||
| KPC-2[6] | F: GCT ACA CCT AGC TCC ACC TTC | 989 | |
| R: ACA GTG GTT GGT AAT CCA TGC | |||
| VIM[7] | F: ATG GTG TTT GGT CGC ATA TC | 500 | |
| R: TGG GCC ATT CAG CCA GAT C | |||
| IMP[8] | F: CAT GGT TTG GTG GTT CTT GT | 270 | |
| R: ATA ATT TGG CFF ACT TTG GC | |||
126株细菌经表型确认有89株为ESBLs阳性, 总阳性检出率71%(89/126). 分别为大肠埃希菌21株、肺炎克雷伯菌50株、变形杆菌18株; 37株其他细菌, 其中ESBLs阴性大肠埃希菌8株、肺炎克雷伯菌2株和变形杆菌1株; 非发酵菌中铜绿假单胞菌7株, 粪肠球菌9株, 白色念珠菌10株.
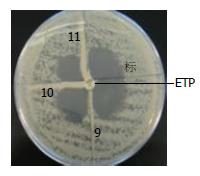
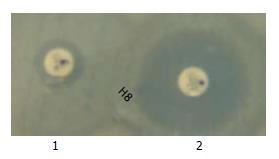
改良Hodge实验中, 3株肺炎克雷伯菌呈现阳性结果(图1), 经PCR扩增、测序, 在GenBank中比对后, 均为KPC-2型耐药基因; 金属酶表型检测结果, 有22株细菌可以被EDTA抑制(图2), 经PCR扩增、测序后证实其为19株VIM型和3株IMP型产金属酶细菌.
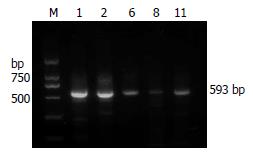
2.3.1 CTX-M型耐药基因: 肺炎克雷伯菌中含CTX耐药基因的菌株占84%(42/50); 大肠埃希菌中为100%(21/21); 而变形杆菌中只有40%(7/18)(表2). 测序结果经GenBank中比对后的分型情况详见表2. PCR结果见图3.
| 亚型种类 | n | 检出率(%) |
| TEM-1b | 68 | 76.4 |
| CTX-M1 | 31 | 34.8 |
| CTX-M9 | 33 | 37.1 |
| SHV-11 | 14 | 15.7 |
| SHV-36 | 7 | 7.9 |
| KPC-2 | 3 | 3.4 |
| VIM | 19 | 21.3 |
| IMP | 3 | 3.4 |
| 新类型 | 1 | 1.1 |
2.3.2 TEM型耐药基因: 肺炎克雷伯菌中含TEM耐药基因的菌株占76%(38/50); 大肠埃希菌为82%(17/21); 而变形杆菌中所有的菌株均含有此耐药基因. 经测序为TEM-1b型耐药基因(表2).
2.3.3 SHV型耐药基因: 肺炎克雷伯菌中只含SHV耐药基因的菌株占14%(7/50); 大肠埃希菌中为83%(17/21); 而变形杆菌中无此耐药基因. 测序为SHV-11型、SHV-36型(表2).
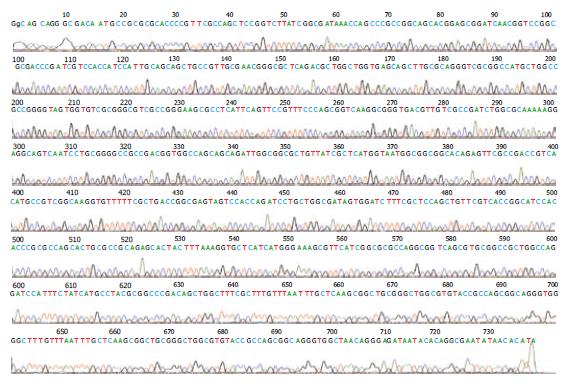
以SHV型引物为例, 测定结果为SHV-11型, 除此之外, 还有SHV-36型耐药基因检出(图4).
TEM型耐药基因全部为TEM-1b型; CTX耐药基因为CTX-M1、CTX-M9型. 测序结果显示碱基发生一些变异, 碱基的缺失有20株、插入的有36株、变异的有33株, 且不同的耐药基因表现出一定的规律性. TEM型耐药基因没有碱基插入现象, 碱基的缺失和变异常常表现在序列末端; CTX型耐药基因没有出现碱基缺失, 而碱基的插入和变异出现在序列前端; SHV型耐药基因中碱基的缺失表现在序列前端、碱基的插入出现在序列末端, 没有出现变异情况.
众所周知, 肠杆菌科及其相关细菌大肠埃希菌、肺炎克雷伯菌、变形杆菌、柠檬酸杆菌、摩根摩根菌、普罗维登斯菌和假单胞菌等, 对人类是极其有益并组成人类肠道的正常菌群[9]. 但是近年来, 有关该类细菌引起的院内感染、尤其是产ESBLs的多重耐药的肠杆菌科细菌引起的内源性感染, 在世界各地陆续有报道, 分离率各不相同. 我国深圳、广东等经济发达地区ESBLs检出率24%-37%[10,11], 姜森等报道的大肠埃希菌ESBLs检出率为40%[12], 台湾地区也报道了有关ESBLs在阴沟肠杆菌中的流行[13]. 本研究126株肠道杆菌ESBLs检出率为71%, 远远高于我国其他地区, 亦高于国外报道[14,15]. 该类患者的共同特点是: 抗生素使用时间大多超过1 wk以上, 且均使用过头孢菌素类、喹诺酮类、碳青霉烯类等两种以上抗菌药物. 考虑与抗菌药物的使用有着密切关系; 而这类腹泻患者患者培养结果大多显示菌群失调, 极易引起内源性感染甚至是院内感染的暴发流行.
ESBLs出现对临床感染治疗带来了极大的威胁, 成为目前亟待解决的问题. 本实验结果显示, 我院因应用抗菌药物导致的腹泻患者大便培养肠道杆菌的ESBLs基因型是以CTX、TEM为主, 大肠埃希菌中100%含有CTX耐药基因, 肺炎克雷伯菌CTX为84%, 变形杆菌100%含有TEM-1b型耐药基因. 与国内报道大致相同[16-18], 与文献报道的亚洲地区流行亦较符合[19,20]. 这类细菌以水解头孢噻肟、头孢曲松等三代头孢菌素为主, 因此对其显示高度耐药性. SHV耐药基因在变形杆菌中没有检出, 肺炎克雷伯菌中检出率为14%, 但在大肠埃希菌中83%的菌株均含有此耐药基因, 该基因是巯基变量(sulphydryl variable)的简称, 以能够水解头孢噻吩中的巯基而得名. SHV型ESBLs是由广谱酶SHV-1的编码基因的1-4个核苷酸突变引起氨基酸改变而形成的一系列酶蛋白, 其中第238位和第240位氨基酸是影响酶水解能力的主要位点. 第238位由丝氨酸替换甘氨酸, 引起对头孢他啶水解能力增强; 第240位由赖氨酸替换谷氨酰胺,造成对头孢噻肟的水解能力增强[21]. 临床表现对头孢他啶、头孢噻肟、头孢哌酮等抗菌药物表现高度耐药性; 但上述三种基因对含酶抑制剂复合制剂敏感性较好、目前且没有发现耐碳青霉烯类抗菌药物菌株. 本组资料显示, 89株产ESBLs耐药菌株往往同时含有多个耐药基因型, 例如: CTX型、TEM型、SHV型可同时出现在同一菌株中, 因此造成了严重的多重耐药的产生, 这一点与王贺等[22]、张晓梅等[23]关于肠杆菌科细菌的研究一致. 从相同耐药基因的高度同源性来看, 他们来源于同一细菌. 又因个别碱基的差异, 可能是导致不同菌株的药敏实验结果不同的主要原因. 有关变异碱基是否导致蛋白表达的变异还有待于我们进一步研究.
本研究还检出产碳青霉烯酶KPC-2型和金属酶IMP和VIM型细菌. KPC-2型是1998年美国首次在肺炎克雷伯菌中检出[24], 以后世界各地关于KPC型耐药菌的报道逐渐增多, 希腊的一家医院内爆发了KPC-2型耐药基因在肺炎克雷伯菌中的流行[25]. 国内山东、江苏等陆续分离出KPC-2型的沙雷菌[26]和大肠埃希菌[27], 河南也曾报道分离出1株产KPC-2的肺炎克雷伯菌[28], 金属酶早在20世纪90年代中国香港在亚胺培南耐药的不动杆菌中就发现IMP-4型耐药基因[29]. 2001年广州报道在绿脓杆菌中发现了IMP-4型耐药基因[30], 浙江大学附属第二医院发现1种IMP-4型产酸肺炎克雷伯菌[31]. 本次我们从大便中检出1株产IMP-4型肺炎克雷伯菌, 河南尚属首例. 这些菌株能够水解碳青霉烯类抗生素导致耐药. 由于其耐药基因存在于质粒上, 且能横向传播, 极易造成医院内感染的暴发流行, 应该引起我们的高度重视. 同时提示, 临床医生在治疗抗生素相关性腹泻时, 应注意防止自身肠源性感染, 慎重使用广谱抗生素; 必要情况下, 可以加服一些肠道微生态制剂[32].
近年来, 由于头孢菌素类抗菌药物的大量使用甚至是滥用, 导致肠道杆菌产超广谱β-内酰胺酶(ESBL)的分离率越来越高, 抗生素相关性腹泻(AAD)在临床发生率亦越来越高. 研究该类患者肠道杆菌产超广谱β-内酰胺酶基因型对于临床抗感染治疗, 预防抗生素相关性腹泻, 控制院内感染的暴发流行有着重要意义.
李瑜元, 教授, 广州市第一人民医院内科.
Beaugerie等对抗生素性腹泻的主要原因进行全面分析, 证实他们多是由梭状芽孢杆菌、金黄色葡萄球菌等引起; 钱英等曾对健康人群肠道大肠埃希菌对氟喹诺酮药物的耐药性及耐药机制进行研究, 证实了我国健康人群肠道大肠埃希菌对氟喹诺酮类抗菌药物的耐药率为很高, 耐药基因主要由gyrA基因和parC基因位点突变造成, 以gyrA基因为主.
本文通过对该类腹泻患者肠道杆菌产超广谱β-内酰胺酶进行基因分型的调查与研究, 不仅发现常见流行的基因型(CTX、SHV、TEM等), 亦发现了新的少见耐药基因(KPC-2、IMP), 这种耐碳氢霉烯类耐药基因的出现会给临床抗感染治疗带来更得大困惑.
超广谱β-内酰胺酶(ESBLs): 属质粒介导, 是当前细菌出现的新的耐药趋势之一, 多发生在肠杆菌科等革兰阴性杆菌中; 细菌一旦产生该类酶, 则对第三代头孢菌素(如头孢噻肟、头孢他啶、头孢哌酮、头孢曲松等)以及单环酰胺类抗生素产生耐药.
本研究创新性强, 方法较先进, 具有较好的学术价值.
| 1. | 罗 学娅, 伦 永志, 高 卫, 胡 海北, 王 强, 刘 莹, 尹 建石, 姜 苏娟. 嗜酸性乳杆菌培养上清液对抗生素相关性腹泻小鼠肠道. 世界华人消化杂志. 2006;14:1870-1873. [DOI] |
| 2. | Kliebe C, Nies BA, Meyer JF, Tolxdorff-Neutzling RM, Wiedemann B. Evolution of plasmid-coded resistance to broad-spectrum cephalosporins. Antimicrob Agents Chemother. 1985;28:302-307. [PubMed] |
| 3. | Clinical and Laboratory Standards Institute. Performance Standards for Antimicrobial Susceptibility Testing. Nineteenth Informational Supplement 2009; 29: M100-S19, Wayne, PA, United States. . |
| 4. | Pagani L, Dell'Amico E, Migliavacca R, D'Andrea MM, Giacobone E, Amicosante G, Romero E, Rossolini GM. Multiple CTX-M-type extended-spectrum beta-lactamases in nosocomial isolates of Enterobacteriaceae from a hospital in northern Italy. J Clin Microbiol. 2003;41:4264-4269. [PubMed] [DOI] |
| 5. | Schlesinger J, Navon-Venezia S, Chmelnitsky I, Hammer-Münz O, Leavitt A, Gold HS, Schwaber MJ, Carmeli Y. Extended-spectrum beta-lactamases among Enterobacter isolates obtained in Tel Aviv, Israel. Antimicrob Agents Chemother. 2005;49:1150-1156. [PubMed] [DOI] |
| 9. | Batchoun RG, Swedan SF, Shurman AM. Extended Spectrum beta-Lactamases among Gram-Negative Bacterial Isolates from Clinical Specimens in Three Major Hospitals in Northern Jordan. Int J Microbiol. 2009;2009:513874. [PubMed] |
| 13. | Yang JL, Wang JT, Lauderdale TL, Chang SC. Prevalence of extended-spectrum beta-lactamases in Enterobacter cloacae in Taiwan and comparison of 3 phenotypic confirmatory methods for detecting extended-spectrum beta-lactamase production. J Microbiol Immunol Infect. 2009;42:310-316. [PubMed] |
| 14. | Dzierzanowska D, Kamińska W, Semczuk K, Borowiec D, Matysiak M, Szumała-Kakol A, Gierczyński R, Patzer JA. Carriage of genes for various extended-spectrum beta-lactamases: a novel resistance strategy of Klebsiella pneumoniae in Poland. Int J Antimicrob Agents. 2010;35:392-395. [PubMed] [DOI] |
| 15. | Mohanty S, Gaind R, Ranjan R, Deb M. Use of the cefepime-clavulanate ESBL Etest for detection of extended-spectrum beta-lactamases in AmpC co-producing bacteria. J Infect Dev Ctries. 2009;4:24-29. [PubMed] |
| 16. | 王 辉, 吴 伟元, 陈 民钧. 肠杆菌科细菌中产超广谱β-内酰胺酶(ESBL)的研究. 中华微生物和免疫学杂志. 2001;21:676-679. |
| 18. | 徐 灵彬, 刘 原, 王 香玲. 西安地区肠杆菌科细菌的产超广谱β-内酰胺酶基因型的研究. 西安交通大学学报(医学版). 2008;29:443-445. |
| 19. | Wu TL, Chia JH, Su LH, Chu C, Kuo AJ, Chiu CH. Dissemination of extended-spectrum beta-lactamase-producing Enterobacteriaceae in intensive care units of a medical center in Taiwan. Microb Drug Resist. 2006;12:203-209. [PubMed] [DOI] |
| 20. | Ensor VM, Shahid M, Evans JT, Hawkey PM. Occurrence, prevalence and genetic environment of CTX-M beta-lactamases in Enterobacteriaceae from Indian hospitals. J Antimicrob Chemother. 2006;58:1260-1263. [PubMed] [DOI] |
| 22. | 王 贺, 杨 启文, 徐 英春, 胡 云建, 孙 景勇, 孔 海深, 吴 伟元, 杨 银梅, 郭 世辉, 朱 震宏. 产超广谱β-内酰胺酶肠杆菌科对头孢美唑等抗菌药物的体外敏感性分析. 中华医院感染学杂志. 2008;18:850-854. |
| 24. | Smith Moland E, Hanson ND, Herrera VL, Black JA, Lockhart TJ, Hossain A, Johnson JA, Goering RV, Thomson KS. Plasmid-mediated, carbapenem-hydrolysing beta-lactamase, KPC-2, in Klebsiella pneumoniae isolates. J Antimicrob Chemother. 2003;51:711-714. [PubMed] [DOI] |
| 25. | Maltezou HC, Giakkoupi P, Maragos A, Bolikas M, Raftopoulos V, Papahatzaki H, Vrouhos G, Liakou V, Vatopoulos AC. Outbreak of infections due to KPC-2-producing Klebsiella pneumoniae in a hospital in Crete (Greece). J Infect. 2009;58:213-219. [PubMed] [DOI] |
| 29. | Chu YW, Afzal-Shah M, Houang ET, Palepou MI, Lyon DJ, Woodford N, Livermore DM. IMP-4, a novel metallo-beta-lactamase from nosocomial Acinetobacter spp. collected in Hong Kong between 1994 and 1998. Antimicrob AgentsChemother. 2001;45:710-714. [PubMed] [DOI] |
| 30. | Hawkey PM, Xiong J, Ye H, Li H, M'Zali FH. Occurrence of a new metallo-beta-lactamase IMP-4 carried on a conjugative plasmid in Citrobacter youngae from the People's Republic of China. FEMS Microbiol Lett. 2001;194:53-57. [PubMed] |
| 31. | Chen LR, Zhou HW, Cai JC, Zhang R, Chen GX. Combination of IMP-4 metallo-beta-lactamase production and porin deficiency causes carbapenem resistance in a Klebsiella oxytoca clinical isolate. Diagn Microbiol Infect Dis. 2009;65:163-167. [PubMed] [DOI] |
编辑: 李瑞敏 电编:吴鹏朕